La biologie synthétique
Définition
Voici la définition de la biologie synthétique trouvée sur Wikipedia :
"La biologie synthétique est un domaine scientifique combinant biologie et principes d'ingénierie dans le but de concevoir et construire ("synthétiser") de nouveaux systèmes et fonctions biologiques." 1
Deux mots clés sont importants dans cette définition. Ce sont les notions de construction et de fonction mais il manque, il me semble, un autre concept important : celui de contrôle d'où la définition que je propose ci-dessous.
Biologie synthétique: construire et donc contrôler, à l'échelle moléculaire, un organisme vivant ou une fonction biologique donnée.
Ces notions de fonction, de construction et de contrôle cachent en réalité une quatrième notion que les scientifiques n'aiment pas trop : la finalité, le but, la téléologie. Ces buts peuvent être par exemple:
- gagner de l'argent (commerce)
- produire une molécule à visée thérapeutique
- produire du bio-fuel
- créer des bio-senseurs qui détectent puis détruisent des cellules cancéreuses
- augmenter l'espérance de vie
- tuer la mort
- créer le surhomme.
Vous voyez que les finalités possibles associées à la biologie synthétique sont nombreuses et il ne faut pas, il me semble, limiter la finalité de la biologie synthétique au commerce comme le fait la "Royale Academy of Enginneering" aux Royaumes Unis :
"The ultimate goal of synthetic biology is to develop commercial applications that will benefit society, i.e to design and build engineered biological systems that process information, manipulate chemicals, fabricate materials and structures, produce energy, provide food, and maintain and enhance human health and our environment." 2
Soyons clairs, je ne nie pas l'existence et l'intérêt des applications commerciales issues de la biologie synthétique. Cependant, la biologie synthétique englobe également des concepts comme le surhomme qui transcendent largement le commerce. En effet, contrôler "moléculairement" un système vivant c'est potentiellement, à terme, contrôler "moléculairement" l'homme. C'est un fait. Et le danger c'est de vouloir le cacher et surement pas de vouloir le dire. Car le cacher serait le meilleur moyen, pour les chercheurs, de se couper de la société et de perdre sa confiance.
Un autre point important à souligner c'est le changement de paradigme (manière de voir le monde) qu'introduit la biologie synthétique. Dans la biologie normale, les chercheurs font des découvertes et les publient. Ils argumentent leurs résultats, proposent une démonstration censée convaincre leurs pairs. Une preuve solide apparait lorsque le pair réussit à reproduire le résultat ce qui n'est pas, en pratique, forcement simple. En biologie synthétique, les choses se passent différemment : en construisant et en contrôlant votre organisme, votre fonction, vous démontrez en même temps votre compréhension quasi-totale du système. La publication n'a plus pour but d'argumenter, de convaincre d'un résultat mais seulement d'expliquer comment le chercheur s'y est pris. Le biologiste synthétique ne démontre pas, il montre, par exemple, la fluorescence de la bactérie qui oscille. La fonction (les oscillations) est démontrée (montrée) d'un simple coup d'oeil: pas de statistiques, pas de graphiques, juste une vidéo. Le résultat est visible et compréhensible par les chercheurs de n'importe quel paradigme (voir la video 6). Prenons une image : si un architecte construit un gratte-ciel : une fois construit et inauguré, il serait fou de remettre en cause son existence ou la réussite de sa construction. En biologie synthétique, c'est la même chose mais le gratte-ciel c'est la souche. La simplicité des démonstrations de la biologie synthétique repose sur l'exécution de la fonction par la souche. Ainsi les démonstrations de biologie synthétique sont très pures par rapport à celles de la biologie classique3 car elles sont basées sur la construction et le contrôle. Et on ne comprend vraiment que ce que l'on peut construire et contrôler. La contrepartie que le chercheur paye pour pouvoir disposer de démonstrations limpides c'est l'énorme difficulté que représente le contrôle moléculaire d'un système vivant à partir de sa séquence ADN.
Construire un système vivant
La biologie synthétique peut être regardée par le prisme des technologies nécessaires pour construire un système vivant de novo. Un jour peut être, nous mettrons des atomes C, H, O, N, P, S etc... dans le réservoir d'une grosse machine reliée à un ordinateur dans lequel nous rentrerons la séquence ADN d'E. coli. Puis on appuiera sur "enter", on attendra quelques minutes puis on récupérera la bactérie créée par la machine en suivant les instructions de la séquence ADN et en utilisant les briques atomiques de base. Mais nous en sommes encore très loin. Voici 4 directions de recherche différentes et complémentaires qui permettront, peut être, d'aboutir un jour à la construction d'un organisme de novo.
- Travailler avec le génome le plus petit possible. Une équipe au Japon cherche le génome minimal c'est-à-dire le génome le plus petit possible capable de coder une entité auto-réplicative en mesure d'utiliser de l'énergie. Ils ont déjà réussi à supprimer plus de 30% du génome de E. coli soit plus de 1300 gènes (Hashimoto et al. 2005; Posfai et al. 2006). D'autres chercheurs travaillent à partir de bactéries possédant un génome encore plus simple (Mycoplasma genitalium) de 580 000 pb soit 7 fois plus petit qu'E. coli (Glass et al. 2006). Cependant cette bactérie ne peut pas vivre seule dans un milieu "classique" : c'est une bactérie parasite qui vit dans des cellules épithéliales. Elle utilise donc les gènes de son hôte de manière indirecte pour vivre.
- Construire un châssis cellulaire. Certains chercheurs essaient de fabriquer des "protocellules". Pour cela, ils fabriquent des vésicules lipidiques qui incorporent une "soupe de molécules" de la machinerie globale nécessaire pour transcrire et traduire (acide-aminés, ribonucléotides, ARN polymerase, ribosomes etc...) ainsi qu'un plasmide (ADN) contenant le gène rapporteur GFP. Les chercheurs arrivent à faire exprimer la GFP dans les vésicules lipidiques pendant plus de 4 jours (Noireaux and Libchaber 2004). Ils arrivent même à enchâsser des pores protéiques dans les membranes pour permettre la diffusion de nutriments et améliorer les performances de ces "protocellules". Cependant il ne s'agit pas de vraies cellules synthétiques car ces "protocellules" ne peuvent évidemment pas s'auto-répliquer.
- Fabrication du support du programme : l'ADN synthétique. Les avancées les plus prodigieuses dans ce domaine nous viennent de l'institut de Craig Venter (dans le Maryland et en Californie). Son équipe a réussi à synthétiser un génome entièrement synthétique en suivant les instructions d'une séquence présente sur un ordinateur (Mycoplasma mycoides, ~1 millions de pb). Puis ils ont transplanté ce génome synthétique dans le châssis cellulaire d'une autre bactérie (Mycoplasma capricolum) la transformant en Mycoplasma mycoides (Gibson et al.). Il faut bien comprendre l'énorme avancée conceptuelle que représente cette expérience. Cela prouve de manière irréfutable la théorie selon laquelle il y a découplage entre la machine (le châssis cellulaire) et le programme (l'ADN) dans un système vivant, exactement comme dans une machine de Turing. On savait déjà effectuer depuis longtemps des transplantations de noyaux qui changeaient effectivement l'espèce. Cependant, rien n'empêchait d'imaginer qu'il y ait des éléments dans le noyau autre que l'ADN intervenant dans le processus.
- Programmer le réseau de régulation dans l'ADN. C'est la difficulté majeure (les 3 points précédents posent un certain nombre de problèmes techniques difficiles mais surmontables à court ou moyen terme). La stratégie la plus commune consiste à essayer de "penser" un réseau de régulation génique comme un réseau électronique. Pour cela, les biologistes couplent de "biobricks". Une biobrick est une séquence d'ADN qui a une fonction définie. Par exemple, une séquence promotrice répresseur ou activateur, une porte AND/OR, un terminateur, un "Ribosome Binding Site" d'une force donnée. En couplant ces "biobricks" ensemble, on peut implémenter des petits systèmes pour l'instant inferieurs à 10 gènes (des compteurs, des filtres passe-bande, des interrupteurs, des oscillateurs etc...) qui exercent leur fonction dans une bactérie comme E. coli par exemple. L'idée est d'utiliser le savoir faire de l'ingénierie pour standardiser ces biobricks et faciliter le travail du biologiste. Mais il faudra petit à petit augmenter la taille de ces réseaux et y inclure de plus en plus de boucles de rétroaction. Or on ne sait pas encore comment on peut contrôler un réseau complexe constitué de plusieurs dizaines, centaines ou milliers de gènes. Le biologiste se heurte à "la complexité" et il a besoin du mathématicien pour l'assister ou plutôt pour que ce dernier s'approprie ce problème et apporte des solutions. Il va falloir apprendre à contrôler le bruit et l'évolution pour que ces derniers ne soient plus des problèmes mais plutôt des solutions. Il faudra d'abord comprendre comment la nature s'y prend pour garder le contrôle du réseau et éviter la dépendance sensitive aux conditions initiales puis s'en inspirer. Et il est fort possible qu'il nous faille dépasser le paradigme un peu naïf de l'électronique.

Le "workflow" en biologie synthétique
Conception et modélisation
Le biologiste synthétique doit programmer une fonction dans l'ADN. Pour cela il dispose de séquences dont la fonction est déjà connue. Son rôle est d'agencer ces séquences pour obtenir la fonction souhaitée. Il va, en général, connecter ensemble des promoteurs, des RBS (Ribosome Binding Site), des terminateurs de transcription avec des gènes. Ces gènes peuvent être soit des facteurs de transcription ou des protéines de transduction du signal qui sont des acteurs directs du réseau de régulation, soit des enzymes ou gènes rapporteurs qui peuvent être des "output".
Pour le design/la conception du réseau, le biologiste peut simplement dessiner le schéma du réseau et évaluer intuitivement les conséquences. Cependant, s'il veut gagner en précision quantitative ou s'il y a des boucles de rétroaction qui rendent la dynamique non intuitive, le biologiste peut utiliser la conception assistée par ordinateur (CAO). Pour cela, il construira et résoudra soit un modèle d'équations différentielles ordinaires soit un modèle stochastique en fonction de l'estimation de l'impact du bruit sur le fonctionnement de son réseau.
Je l'ai dit dans le chapitre sur la modélisation : il me semble que le biologiste doit fixer le caractère prédictif du modèle comme priorité absolue. Ce n'est pas toujours le cas : en biologie des systèmes par exemple, il est possible (et utile) de faire de très bons modèles descriptifs mais non prédictifs.
Pour conserver le caractère prédictif: il faut faire des modèles simples mais non simplistes. Il faut limiter à tout prix le nombre de paramètres car la valeur numérique de ces derniers est souvent inconnue. Selon moi (et grossièrement), un modèle non linéaire contenant plus de 10 gènes régulés n'a que très peu de chance de générer des prédictions fiables et donc utilisables. Or la fiabilité des prédictions est cruciale car les étapes de biologie moléculaire qui suivent sont extrêmement difficiles à réaliser en pratique. Si le modèle génère une prédiction fausse, on risque de passer 6 mois à faire de la biologie moléculaire pour rien.
Il n'existe pas encore, à ma connaissance, d'environnements de développement intégrés (IDE) propres à la biologie synthétique. Selon moi, un environnement de ce type devrait associer:
- Une gestion simple et intuitive de la séquence ADN avec des fonctionnalités de couplage de " biobrick" et donc de "clonage in silico". La connexion aisée à des bases de données sur internet permettrait d'importer des objets/de l'information facilement (biobrick, valeur de paramètres etc...)
- Des simulateurs déterministes et stochastiques faciles d'utilisation qui permettraient de prédire la dynamique du système dans des conditions standards.
- Des fonctionnalités (rational design, évolution dirigée) qui assisteraient le biologiste pour " tuner" les paramètres du système.
Les IDE qui s'imposeront seront les plus simples et les plus intuitives. Les "usines à gaz" demandant un savoir trop poussé n'auront aucune chance de séduire les biologistes.
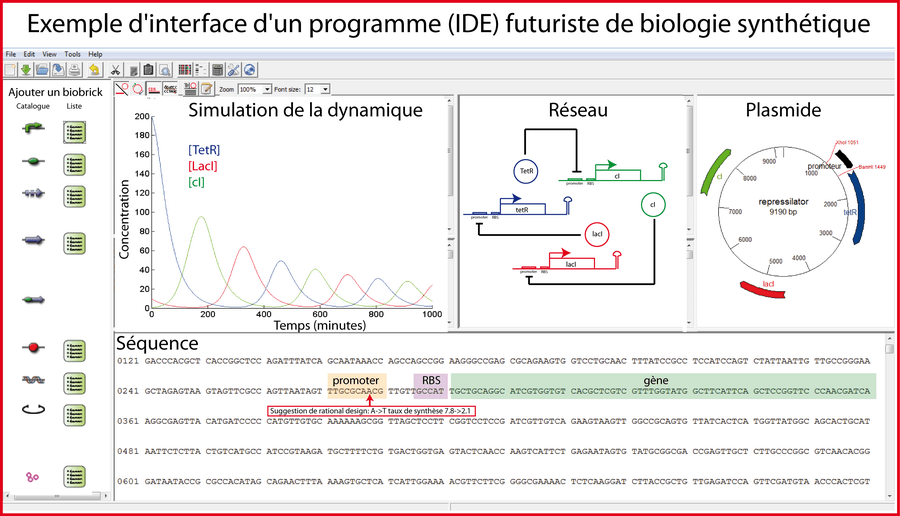
Ecriture du programme ADN
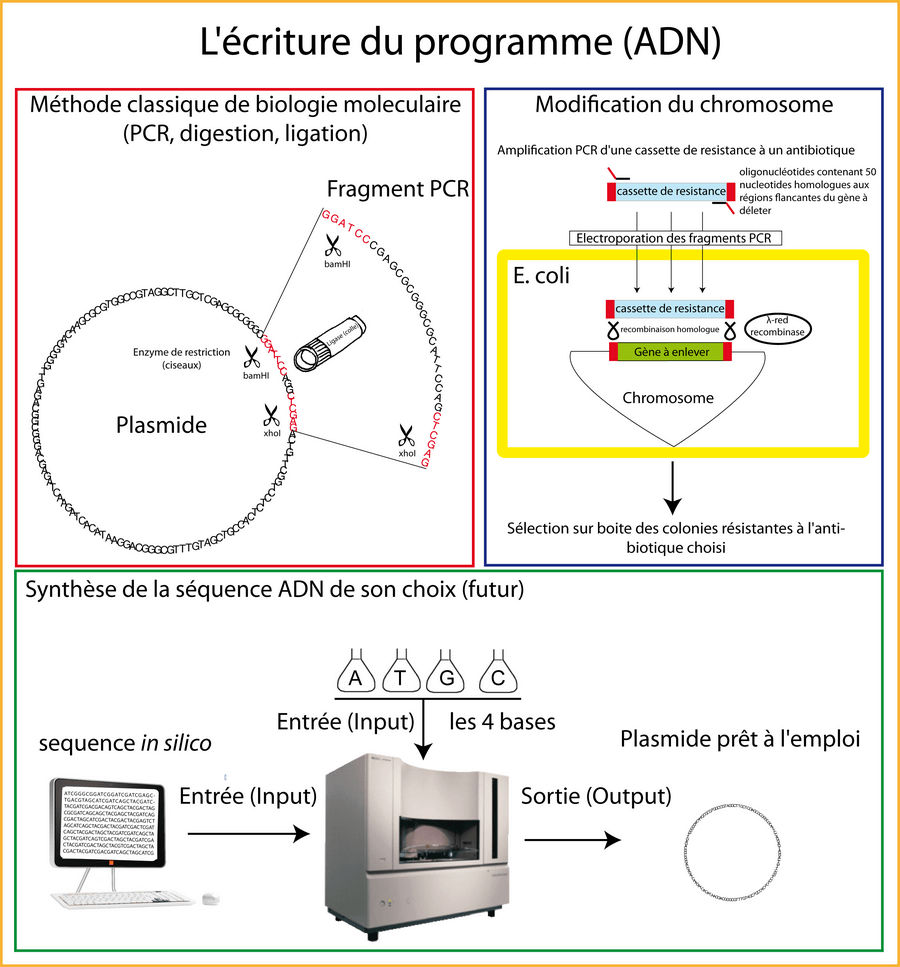
Les techniques classiques de biologie moléculaire s'apparentent au découpage de l'ADN avec des ciseaux très spécifiques (les enzymes de restriction), puis au collage (raboutage) avec de la colle (la ligase). La PCR permet d'amplifier le gène ou la séquence ADN de son choix en rajoutant aux extrémités des petites séquences appelées site de restriction que "les ciseaux" (les enzymes de restriction) reconnaissent. On peut ensuite venir "coller/rabouter/placer" ce fragment ADN contre une autre séquence ADN, un plasmide par exemple. On appelle cela le clonage (rien à voir avec la brebis Dolly). En théorie, les clonages sont simples : il n'y a apparemment aucune raison que cela ne marche pas quand on respecte le protocole. En pratique, je connais peu de gens qui ne considèrent pas le clonage comme une lutte. Ce qui devrait prendre une ou deux semaines prend souvent des mois, au grand désespoir des biologistes. La difficulté de la biologie moléculaire est un des freins au développement de la biologie synthétique. Fabriquer un plasmide avec une fonction donnée coûte très cher, vu le temps nécessaire (et donc le salaire) que le biologiste y consacre. Cependant certains biologistes ont des doigts de fées. Ils valent chers sur le marché du travail. Pour ma part, force est de constater que la biologie moléculaire ne m'a pas dans ses petits papiers.
Une autre technique consiste à supprimer les étapes de digestion et de ligation en les remplaçant par la technique de recombinaison homologue (crossing over). Cette technique permet de modifier un plasmide mais également d'effectuer directement des modifications sur le chromosome bactérien (Datsenko and Wanner 2000; Yu et al. 2000).
Enfin un grand espoir repose également sur la synthèse de novo de gènes (ou de n'importe quelle séquence ADN). Cette technique devient, avec le temps, de plus en plus rapide et fiable et de moins en moins onéreuse. L'idée est simple : alléger la charge de travail du biologiste synthétique futur. Dans l'idéal, ce dernier ne devrait se concentrer que sur la partie conception et caractérisation et sous traiter la partie synthèse/écriture du programme à une entreprise. Après la phase de conception, le biologiste n'aurait plus qu'à envoyer la séquence "designée" de son plasmide à une entreprise qui se chargerait de le synthétiser.
Nous avons vu la partie "écriture" de l'ADN. Notez que la partie "lecture" est également essentielle en biologie synthétique. C'est la technologie du séquençage. Les prix et performances de la "lecture" et de "l'écriture" c'est-à-dire du séquençage et de la synthèse s'améliorent de façon exponentielle avec le temps. Le développement de ces technologies devrait pouvoir accélérer le développement de la biologie synthétique.
La caractérisation
La caractérisation consiste à évaluer/exécuter le programme ADN. On place ce programme ADN dans un châssis bactérien et on place la bactérie dans un milieu particulier à définir. Puis on regarde si l'exécution du programme permet d'obtenir la fonction désirée (le phénotype). Ainsi la caractérisation du programme ADN se fait toujours in vivo. Pour évaluer la fonction, les biologistes placent sur l'ADN des gènes rapporteurs qui fournissent une sortie (output). Il peut s'agir de la GFP (fluorescence), de la luciferase (luminescence) ou plus généralement d'enzymes qui catalysent une réaction chimique facilement quantifiable ce qui permet de remonter à la concentration de la dite enzyme (β-galactosidase, gènes de résistance aux antibiotiques etc...).
Les outils/appareils les plus classiques pour évaluer l'exécution du programme
- le lecteur de microplaque pour une caractérisation à l'échelle de la population. Les puits d'une microplaque contiennent du milieu avec les bactéries. Le lecteur de microplaque peut mesurer des paramètres comme l'absorbance, la fluorescence ou la luminescence. Ce type de données peut facilement être comparé aux solutions d'un modèle d'équations différentielles ordinaires. Lors d'une cinétique d'expression génique, un lecteur de microplaque peut faire des mesures toutes les minutes (par exemple) ce qui permet d'obtenir des données d'expression avec une haute résolution temporelle.
- Le cytomètre en flux fournit des données sur chaque bactérie. Par exemple, la taille (l'aire), ou la fluorescence. Ces données permettent d'étudier la distribution de fluorescence des cellules ou la distribution des aires. Elles sont donc particulièrement propices à l'évaluation des prédictions générées par un modèle stochastique.
- La microscopie à fluorescence. Elle permet de générer des images ou des films (cinétiques) d'expression génique à l'échelle de la cellule isolée. Une seule image peut contenir plusieurs types d'informations en jouant sur la superposition des couleurs: par exemple, on peut générer une image contenant l'information du contraste de phase, l'information de la fluorescence verte pour "monitorer" la concentration de la GFP et l'information de la fluorescence rouge pour "monitorer" la concentration de la RFP. Des logiciels très puissants (technologie scannR chez Olympus) permettent de détecter et comptabiliser en quelques secondes les paramètres (aire, fluorescence...) de milliers de cellules isolées à partir des images. Cela permet, comme pour le cytomètre en flux, de générer des graphes de dispersion (scatter plot) ou des distributions mais avec, en plus, l'énorme avantage de pouvoir vérifier ses résultats en regardant directement les images des cellules.
- La microfluidique : couplée à la microscopie à fluorescence, la microfluidique est un domaine très prometteur pour effectuer de nombreuses mesures en parallèle à l'échelle de la cellule isolée.
- Tous les dispositifs expérimentaux que les chercheurs peuvent imaginer : par exemple, un chercheur peut utiliser comme "output" la mobilité des bactéries. Il fabrique alors un dispositif expérimental en utilisant une gélose nutritive semi-solide où les bactéries "nagent". En fonction de la séquence ADN programmée, ces dernières nagent plus ou moins vite. Il suffit alors de mesurer la distance qu'elles parcourent et de s'en servir comme données reflétant/rapportant l'expression de tel ou tel gène nécessaire à la mobilité.

Optimisation de ces 3 étapes
Pour accélérer le développement de la biologie synthétique, chacune de ces 3 phases (conception, écriture et caractérisation) devrait être optimisée, standardisée, si possible automatisée le tout sous forme d'approche haut débit.
Prenons l'exemple du repressilator (voir le repressilator dans le chapitre sur la modélisation). Ce plasmide code un système qui génère des oscillations dans la concentration des protéines. Il est composé de 3 promoteurs et de 3 gènes. S'il fallait recommencer sa construction en privilégiant une approche haut-débit optimisée, voilà comment je m'y prendrais.
Utilisation de l'IDE futuriste décrite ci-dessus. Le biologiste ajoute les "biobrick" par glisser/déposer ce qui crée le squelette de base du plasmide. Puis un algorithme de simulation calcule la dynamique du système. Le logiciel assiste ensuite le biologiste pour détecter les nucléotides qui sont les points chauds critiques influant sur les paramètres du système. Ce sont les nucléotides des promoteurs, des RBS etc... qui modulent les taux de synthèse, l'affinité des facteurs de transcription sur les promoteurs, etc... Une fois une dizaine de points chauds choisis par l'utilisateur, le logiciel génère toutes les combinaisons de séquences possibles. Par exemple, une centaine. L'algorithme de simulation évalue à nouveau ces 100 séquences et met éventuellement de coté les séquences qui n'auraient aucune chance de générer le phénotype voulu. Puis l'expérimentateur envoie de manière semi-automatisée (gestion par le logiciel) les séquences à une entreprise pour synthèse. Cette dernière renvoie une microplaque contenant soit les 100 plasmides, soit les 100 souches bactériennes contenant les dits plasmides. Le biologiste peut alors caractériser les 100 séquences ADN avec un dispositif microfluidique contenant le nombre de canaux nécessaires pour tester les 100 souches en une seule cinétique (en parallèle). Il suffit ensuite d'identifier les souches dont la concentration de la GFP oscille à la fréquence et l'amplitude souhaitée.
Au jour d'aujourd'hui, ma prédiction est qu'il faut plusieurs années (entre 3 et 10 ans) à une équipe de 3--5 personnes pour designer, construire et caractériser ces 100 plasmides à la main sans même avoir l'assurance d'obtenir, à la fin du temps imparti, un plasmide "parfait" c'est à dire générant des oscillations à telle fréquence et de telle amplitude par exemple. Ce qu'il faut c'est donc travailler avec des ingénieurs/thésards/chercheurs à l'optimisation du workflow décrit ci-dessus de telle manière à réduire les temps à un niveau acceptable : quelques mois maximum pour caractériser le phénotype de 100 souches contenant 100 programmes bien définis.
Le charbon
L'IGEM (International Genetically Engineered Machine compétition) est une compétition internationale de biologie synthétique entre étudiants. Cette compétition, lancée par le MIT en 2004, a pour but de développer la biologie synthétique. Elle compte, en 2011, 164 équipes soit plus de 1000 étudiants.

La construction de souches4 réalisant des fonctions plus ou moins loufoques est confiée aux étudiants IGEM5 pour plusieurs raisons :
- La biologie synthétique représente le futur / la science-fiction et les jeunes ne s'y trompent pas (un peu comme l'informatique et la programmation dans les années 70--80).
- Cette compétition est une excellente publicité pour le développement de la biologie synthétique
- il faut de l'imagination et ne pas être trop formaté pour imaginer de nouvelles souches, de nouvelles fonctions. En cela les étudiants sont très précieux car ils apportent de nouvelles idées.
- la biologie moléculaire reste encore très difficile. L'acharnement nécessaire est confié à la jeunesse.
Certains pourraient croire que la biologie synthétique est confiée aux étudiants car elle relève de l'ingénierie et non de la science. On pourrait penser qu'il s'agit simplement de l'application de protocoles standards, relativement simples, pour "jouer" à fabriquer des souches loufoques. On pourrait croire également que les chercheurs, ayant déjà largement déblayé le terrain, auraient confié la tache de poursuivre la biologie synthétique à des ingénieurs pour que ces mêmes chercheurs puissent se focaliser sur des choses plus utiles ou plus abstraites. Cette manière de pensée potentielle ne reflèterait pas selon moi la réalité. La biologie synthétique ne correspond pas à un transfert de compétence des chercheurs aux ingénieurs. La biologie synthétique amorce un changement de paradigme.
L'ancien paradigme ou cadre de recherche du 20ème siècle fixait comme objectif la découverte de nouvelles entités (ADN, protéine, ARN) ou de nouveaux mécanismes ou fonctions de protéines. La dernière grande découverte de ce type a marqué la fin du 20ème siècle : la découverte de l'ARN interférence. Le nouveau paradigme, celui du 21ème siècle, c'est celui du système, du réseau, de la complexité et de d'information. Maintenant que l'on connait presque tous les éléments de la cellule, il faut faire le chemin inverse et essayer de remettre ces éléments ensemble en reconstruisant le réseau. L'objectif ce n'est plus la découverte de nouvelles entités ou de nouveaux mécanismes, l'objectif c'est le contrôle du système. Ce contrôle fournit une preuve de qualité supérieure de la compréhension d'un système biologique. Et bien caché derrière le contrôle se cache bien sûr (je le répète) la notion de finalité c'est-à-dire la téléologie.
Le but caché derrière la construction d'une souche loufoque, c'est la construction et le contrôle de souches de plus en plus complexes : c'est-à-dire accroitre les capacités computationnelles d'un système vivant (le wetware). Les progrès futurs qui vont changer nos vies résident dans l'accroissement du contrôle de ces capacités computationnelles et surement pas dans la découverte d'une énième entité ou d'un énième mécanisme inconnu. Le changement de paradigme est en marche et on sait depuis Thomas Kuhn6 que cela passe souvent par le renouvellement des générations de chercheurs. Les anciens partiront à la retraite7 et les nouveaux seront les étudiants IGEM qui font, je le crois, souvent partie des très bons étudiants : motivés8 , aptes au travail en équipe et prêts à se mobiliser pour un projet qu'ils doivent mener eux-mêmes dés le premier cycle universitaire. Et, vous l'aurez compris, cette génération d'étudiants aura été "élevée" avec les notions de systèmes, de réseaux, de bruit, de complexité, de chaos, d'informations, etc... Ils auront baigné dans la fournaise de l'interdisciplinarité réelle (biologie, physique, mathématique, chimie, informatique etc...). Et c'est dans cette fournaise que sont susceptibles de se faire les découvertes fondamentales qui changeront le plus la vie des hommes.
Listing de quelques fonctions existantes en biologie synthétique
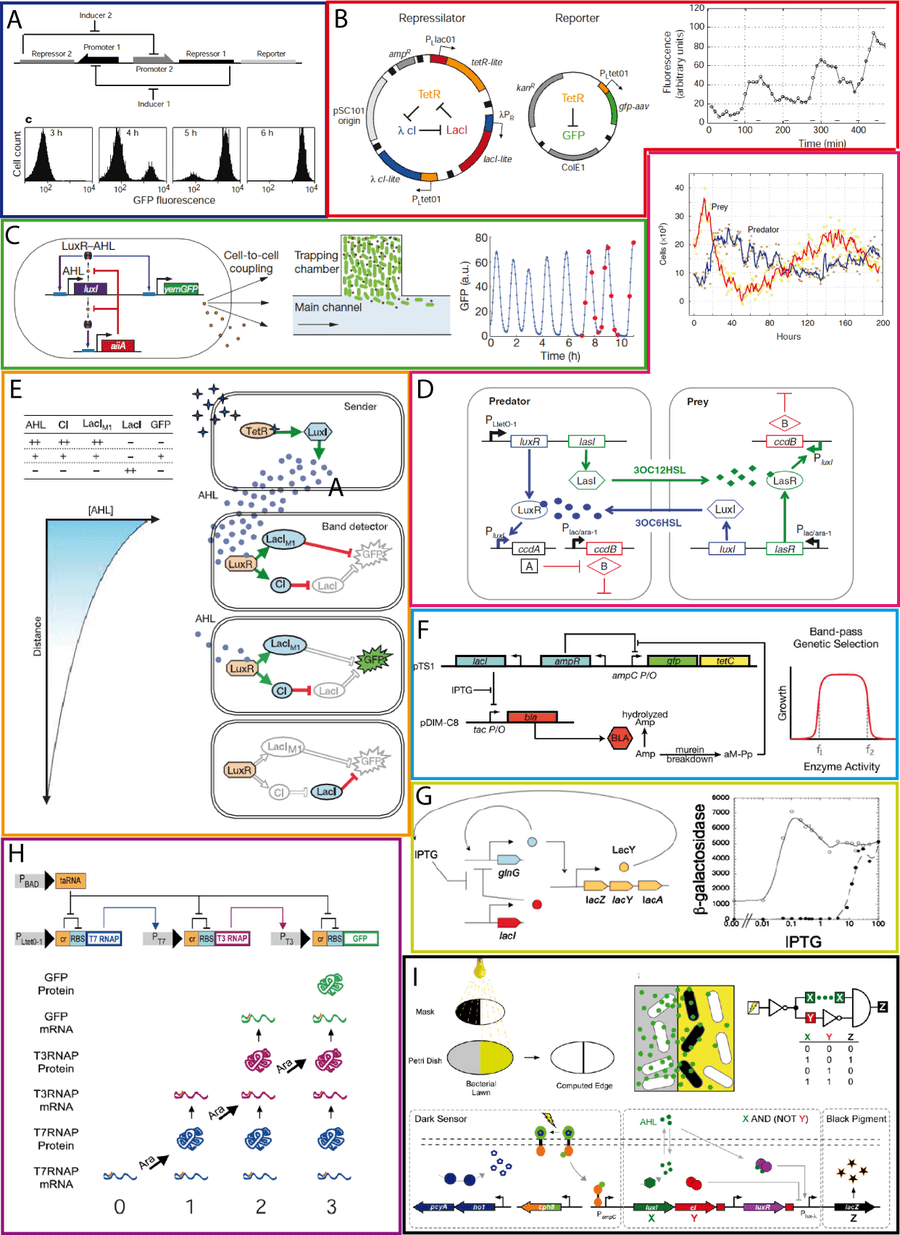
Ma liste n'est pas exhaustive et a principalement pour but de donner des références bibliographiques à ceux que cela intéresserait. La figure 7 fournit quelques schémas extraits des publications citées.
- L'interrupteur génétique (le toggle switch). Cette construction, basée sur une double répression, permet de maintenir "on" l'expression d'un gène et "off" le deuxième et réciproquement (Gardner et al. 2000). Voir figure 7A. Voir le modèle correspondant au toggle switch. Voir le chapitre sur les systèmes dynamiques.
- L'oscillateur (le repressilator). Cette construction, basée sur une triple répression, permet de faire osciller la concentration de 3 protéines répresseurs (Elowitz and Leibler 2000). Voir figure 7B et figure 6. Voir le chapitre sur la modélisation.
- Les oscillateurs multicellulaires. Il s'agit d'un système synthétique incluant une molécule de communication extracellulaire permettant de synchroniser les oscillations à l'échelle de la population (Danino et al. 2010). Voir figure 7C.
- Système oscillant proie--prédateur : Il s'agit d'un système de double communication intercellulaire mettant en jeu 9 gènes. C'est actuellement le système le plus complexe (en terme de nombre de gènes) que je connaisse (Balagadde et al. 2008). Voir figure 7D.
- La programmation de patterns multicellulaires : une bactérie "sender" produit un composé extracellulaire l'AHL (Acyl-Homoserine Lactone) qui est détecté par une bactérie "receiver". Les cellules "receiver" expriment ou non la GFP en fonction de la concentration de l'AHL et donc de la distance qui les séparent des "senders" ce qui forme des jolies patterns (Basu et al. 2005). Voir figure 7E.
- Le filtre passe-bande. L'utilisation d'un "incoherent feed forward loop" permet d'exprimer un gène dans une fourchette de concentration bien précise d'un activateur. Il y a une borne vers le haut et une borne vers le bas (Sohka et al. 2009). Voir figure 7F.
- La mémoire : le couplage de boucles de retroaction positive peut générer un système bistable (phénomène d'hystérèse) pouvant servir de mémoire (étudié dans le chapitre sur la modélisation) (Chang et al. 2009). Voir figure 7G.
- Le compteur : un système synthétique capable de compter jusqu' à 3 événements d'induction (Friedland et al. 2009). Voir figure 7H.
- Détection de frontière : il s'agit d'un programme ADN dans E. coli qui lui permet de détecter et marquer les lignes ou frontières dans une image en secrétant un pigment noir à cette endroit seulement (Tabor et al. 2009). Voir figure 7I.
- Appareil photo : la bactérie Escherichia coli est transformée en un appareil photo de 100 millions de pixel. Un récepteur stimulé par la lumière déclenche une cascade de réaction qui entraine l'expression d'un gène (lacZ) produisant un pigment noir (Levskaya et al. 2005).
Le futur en biologie synthétique
Petit détour par la théorie des graphes
En théorie des graphes, un graphe correspond à un réseau abstrait et n'a donc rien à voir avec un graphique. Un graphe est un ensemble de points (pour nous les gènes) reliés par des arêtes (les interactions). Etant donné que, dans un réseau biologique, ces arêtes sont orientées, on parle de graphe orientés et je parlerai de flèches plutôt que d'arêtes.
Accroitre les capacités computationnelles d'un système biologique synthétique passe par l'augmentation de la taille et de la complexité des graphes. Autrement dit, il faudra réussir à augmenter:
- le nombre de gènes N
- le nombre de flèches F unissant ces gènes.
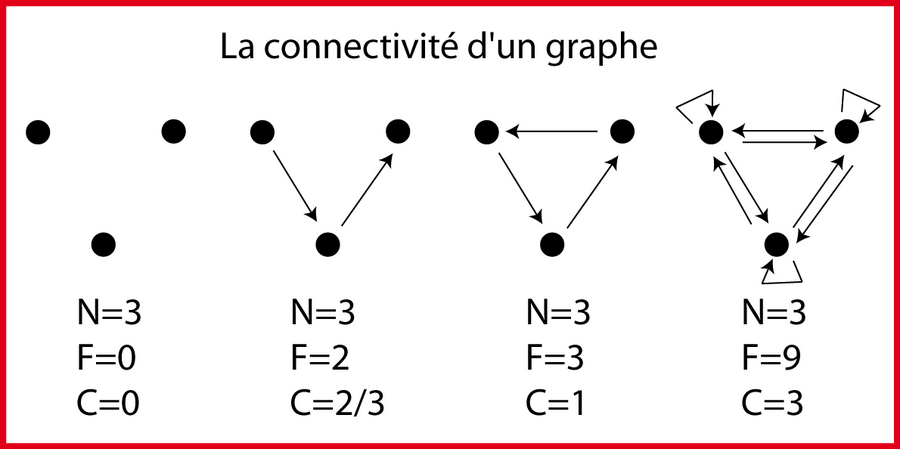
La connectivité C d'un réseau est défini comme le rapport du nombre de flèches sur le nombre de gènes soit C=F/N. Prenons le cas simple où on ne sépare pas l'activation et l'inhibition (autrement dit une flèche peut représenter soit l'une soit l'autre) :
- Si C=0. Il n'y a pas d'interaction et l'expression des gènes de l'organisme est dite constitutive. Par exemple, on peut vouloir faire produire à une souche (usine bactérienne) un métabolite particulier qui nécessite une chaine linéaire de 5 enzymes. L'expression de chacune de ces 5 enzymes est indépendante. Il n'y a pas de régulation spécifique. On place devant chaque gène un promoteur constitutif de la force adéquate de manière à optimiser la production du dit métabolite.
- Si C=F/N= (N-1)/N. Dans ce cas, il y a une flèche de moins que le nombre de gènes. Si chaque gène est connecté au réseau alors la dynamique est parfaitement intuitive car il ne peut pas y avoir de boucles de rétroaction. Cela permet potentiellement de se passer d'un modèle mathématique en ce qui concerne le comportement dynamique du système.
- Si C=1. Il y a autant de flèches que de gènes. Si chaque gène est connecté au réseau, Il existe forcement une boucle de rétroaction rendant potentiellement la dynamique du système non intuitive. Un modèle mathématique risque d'être indispensable.
- Si C=N (car (N(N-1)+N)/N=N). Le système contient N(N-1)+N flèches. Dès que le nombre de gènes est supérieur à 2, la dynamique du système est impossible à prédire sans modèle.
Il y a donc deux enjeux en biologie synthétique :
- Augmenter N tout en fixant C < 1. Cela permettra d'apprendre à travailler avec des réseaux de grande taille, par exemple contenant plus de 10, 20, 50, 100 gènes. La dynamique restera totalement intuitive. La difficulté est plutôt technique : il faut réussir à contrôler, fixer, optimiser l'expression d'un grand nombre de gènes dans une cellule pour obtenir la fonction souhaitée.
- augmenter C tout en fixant N. La dynamique n'est plus intuitive et il faut absolument un modèle mathématique. La difficulté est conceptuelle : comment augmenter C tout en gardant le contrôle d'un système contenant un grand nombre de boucles de rétroaction ? comment faire pour se prémunir de la dépendance aux conditions initiales ? faut-il utiliser les techniques de l'électronique : modularité, rétroactivité (Del Vecchio et al. 2008; Sauro 2008) ? Ou bien faut-il comprendre comment la nature s'y prend et utiliser sa méthode (évolution ?) ? La nature sait-t-elle d'ailleurs travailler avec des hautes valeurs de C ?
La prochaine génération de réseaux synthétiques
Certaines idées des paragraphes qui suivent se basent sur l'excellente publication Next generation synthetic gene network de Timothy Lu paru en 2010 dans Nature Biotechnology (Lu et al. 2009). Je conseille vivement sa lecture car c'est une niche à bonnes idées. D'ailleurs, certaines de ces idées sont des pistes que je garde en tête pour mes recherches futures. Et je serais donc très heureux de discuter avec un lecteur qui réfléchirait à fouiller en direction de l'une de ces perspectives.
Filtres "tunable"
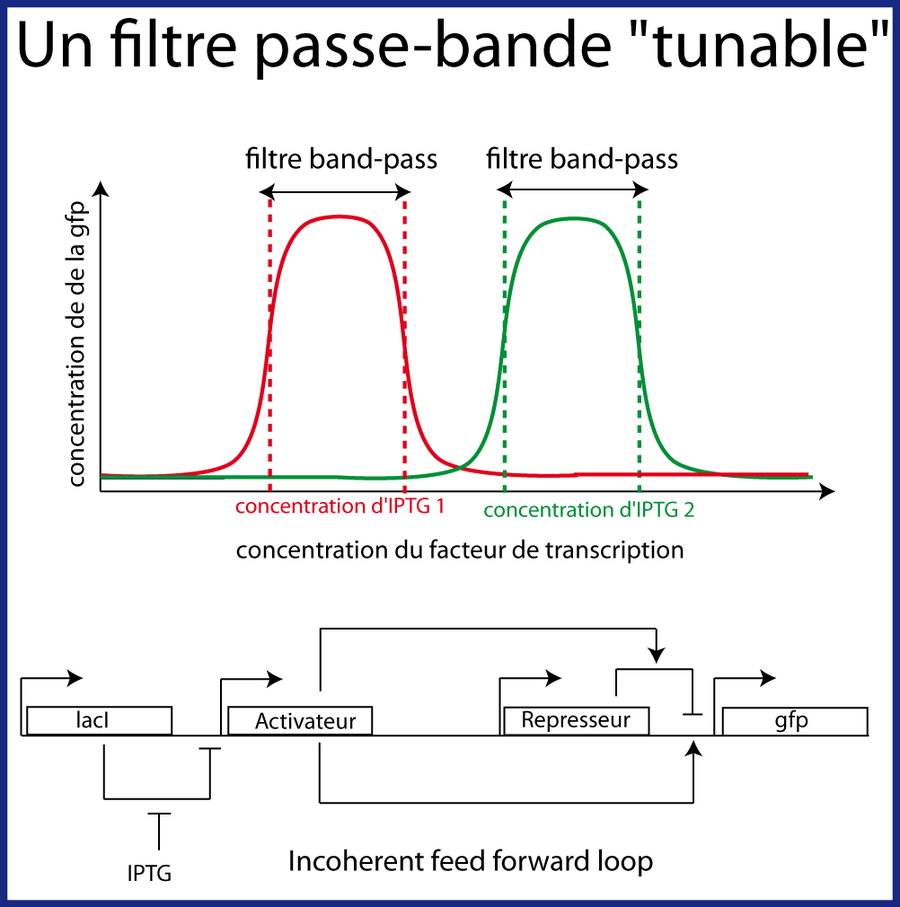
La technique actuelle en biologie synthétique est la suivante : le biologiste connecte des séquences d'ADN entre-elles ("plug and play"), chacune ayant une fonction donnée de manière à créer une séquence nouvelle, plus complexe. Souvent cette séquence ne fournit pas du premier coup la fonction attendue, il faut "tuner" certains paramètres en modifiant soit rationnellement (rational design) certains nucléotides (par exemple pour diminuer la force d'un promoteur) soit en utilisant des techniques d'évolution dirigée (voir chapitre sur l'évolution). Dans les deux cas, il faut venir modifier la séquence ADN. Et que ce soit nous ou l'évolution qui le fasse, cela prend toujours du temps. L'idée c'est donc de créer des composants (des filtres) dont les paramètres sont "tunables" beaucoup plus facilement c'est-à-dire sans avoir à modifier la séquence ADN. C'est le cas par exemple du filtre passe-bande dont j'ai déjà parlé ci-dessus (Sohka, Heins et al. 2009) dont la bande (la zone d'activité) peut être ajustée sur plus de 4 ordres de grandeur en faisant varier un stimulus externe (inducteur).
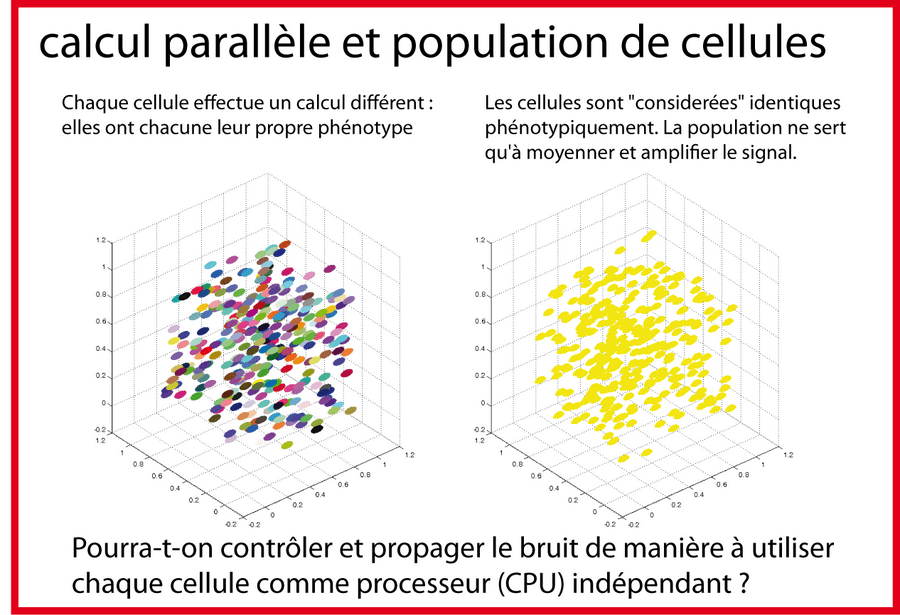
L'utilisation du bruit
Le bruit est souvent considéré comme gênant alors qu'on soupçonne les populations de cellules de l'utiliser pour "computer" des "outputs". L'idée est d'apprendre à utiliser et contrôler le bruit. Il faut pour cela créer des générateurs de bruit "tunables". Le couplage de motifs propageant le bruit pourrait générer une grande diversité dans la population clonale de cellule. Diversité qui pourrait peut-être démultiplier les capacités de calcul du Wetware. Comment ? Je ne sais pas. Mais les capacités de calcul sont souvent réduites/cantonnées au réseau intracellulaire et les millions de cellules dans un puits d'un lecteur de microplaque ne servent qu'à amplifier le signal. Or en générant de la diversité grâce au bruit dans une population clonale, on peut peut-être faire en sorte que chaque cellule résolve un calcul particulier. La population traiterait alors les calculs de manière massivement parallèle. Peut-être que cette question théorique intéressera les mathématiciens / modélisateurs ?
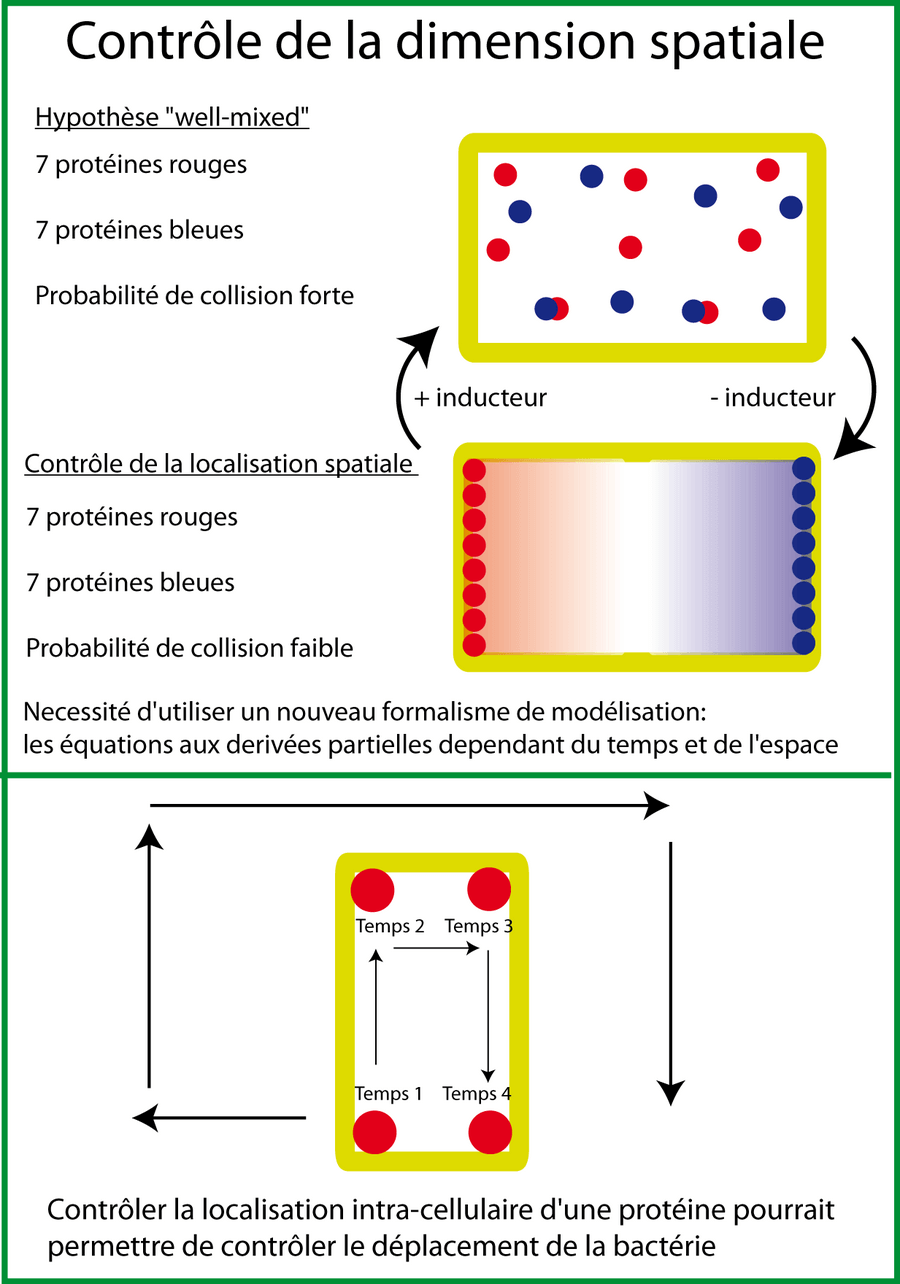
Utilisation de la dimension spatiale
Pour l'instant, il me semble que les systèmes synthétiques sont incapables de contrôler et d'utiliser la position (coordonnée XYZ) d'une entité (par exemple une protéine). Ces systèmes se basent sur l'hypothèse "well mixed" de répartition homogène des protéines et autres molécules dans la cellule. Cependant, cette hypothèse devient faible lorsqu'une protéine est en un seul exemplaire dans la cellule : connaitre sa localisation devient une information de poids. Et Il existe des méthodes de microscopie "at the single molecule level" qui permettent de localiser assez précisément des molécules fluorescentes uniques (Elf et al. 2007; Li and Elf 2009). En contrôlant la localisation, on pourrait faire beaucoup de chose :
- imaginez un facteur de transcription qui serait fixé aux membranes dans une condition donnée. Puis lorsqu'un événement bien précis arriverait, il se détacherait et viendrait se fixer à l'ADN pour activer l'expression de sa cible.
- imaginez que vous arriviez à maintenir deux sous-unités d'une protéine chacune dans un pôle opposé de la cellule jusqu' à ce qu'un événement choisi les fasse lâcher leurs pôles puis se rencontrer en engendrant un phénotype particulier.
On pourrait jouer sur la probabilité de rencontre entre deux molécules en faisant varier la distance spatiale qui les sépare ou en jouant sur l'affinité qu'a une protéine pour une zone/entité particulière. Les modèles d'équations différentielles ordinaires ne suffisent plus, il faut passer à un formalisme utilisant des équations aux dérivées partielles plus adaptées aux modèles spatiaux.
Notez que le contrôle de la dimension spatiale intracellulaire permettrait9 le contrôle spatial extracellulaire. On pourrait ainsi programmer E. coli comme un petit robot se déplaçant aux endroits voulus en fonction des stimuli externes.
Réseau de calcul basé sur les protéines
Nous avons beaucoup discuté de réseaux synthétiques basés sur l'expression des gènes. Ceux-ci ont l'énorme avantage de nous faire bénéficier d'outputs (les sorties) très appréciables : les gènes rapporteurs (GFP, lacZ, luciferase...). Mais le gros défaut des réseaux de gènes, c'est l'échelle de temps longue (minutes, heures) sur laquelle ils opèrent ce qui rend les calculs peu "réactifs". La biologie synthétique devrait regarder en direction de la transduction du signal qui est parfois oubliée ; en effet, l'allostérie (les molécules qui s'emboitent) et les modifications chimiques (phosphorylation, acétylation etc...) se produisent à une échelle de temps beaucoup plus rapide (secondes). Par exemple, construire et contrôler un réseau non linéaire de phosphorylation pourrait permettre la création d'ordinateurs cellulaires très rapides. Chez E. coli, il faut peut être regarder du coté des systèmes à deux composantes. Le problème c'est qu'il nous manque des outputs permettant de contrôler l'état du système in vivo et en temps réel comme c'est le cas avec les gènes rapporteurs. Ce qu'il faudrait, c'est inventer des protéines qui ne "fluorescencent" ou "luminescent" que lorsqu'elles sont phosphorylées par exemple. Puis on créerait des contrôleurs à activité kinase et phosphorylase spécifiques de ces protéines "rapporteurs".
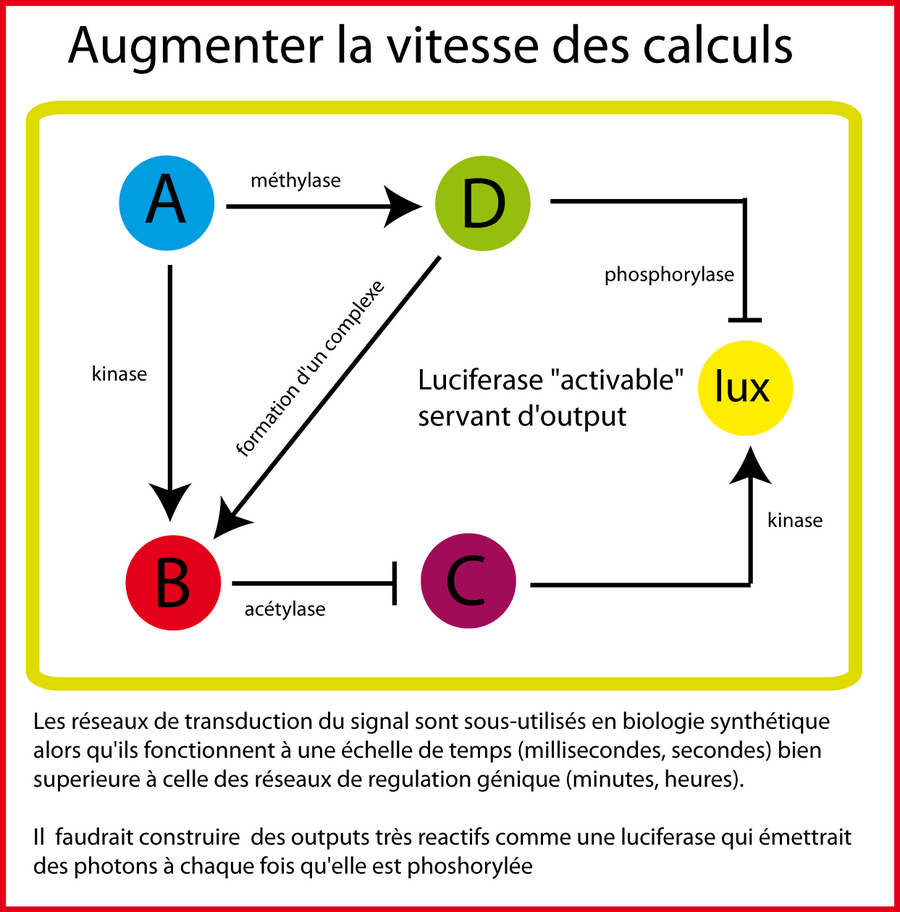
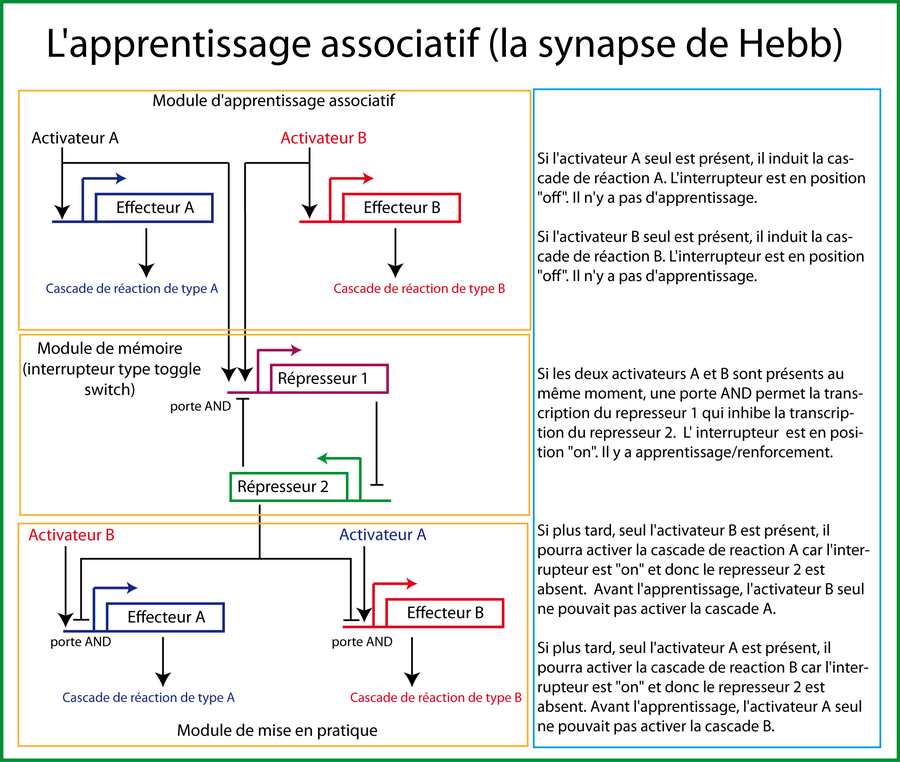
Des réseaux qui apprennent
Si vous êtes biologiste, vous avez peut être entendu parler de la synapse de "Hebb". Ce chercheur a fournit une contribution très importante à la neurobiologie : l'idée que deux neurones en activité au même moment créent ou renforcent leur connexion de sorte que l'activation de l'un par l'autre soit plus facile à l'avenir. C'est ce mécanisme de renforcement à l'échelle du système nerveux qui pourrait expliquer, dans une certaine mesure, les mécanismes de l'apprentissage.
Si vous êtes informaticien ou mathématicien, vous préférez sans doute que je vous parle des réseaux de neurones artificiels qui permettent aux programmes de reconnaissance vocale de s'améliorer au cours du temps par exemple. En effet, un perceptron multicouche apprend à reconnaitre les mots que vous prononcez au fur et à mesure que vous lui parlez. Le programme s'améliore.
Peut-on encoder dans l'ADN un programme/un réseau capable d'apprentissage ? Oui. En suivant les conseils dispensés dans la publication de Lu (Lu et al. 2009). Le cahier des charges est simple : le réseau doit être capable de répondre différemment à un même stimulus en fonction de son apprentissage. Voici le design basic d'un réseau permettant l'apprentissage associatif. La description est un petit peu difficile : aidez vous de la figure 14. Deux facteurs de transcription A et B sont exprimés en réponse à deux stimuli bien distincts. Supposons que chaque facteur de transcription déclenche l'expression de deux protéines effectrice A et B elle-même contrôlant chacune une cascade de réactions A et B. Lorsque les deux stimuli se produisent au même moment : les deux facteurs de transcription sont transcris et ils activent un interrupteur (un toggle switch). Ceci crée la mémoire associative. Plus tard, si un seul des facteurs de transcription (par exemple B) est produit, la logique de la porte AND entre le toggle switch et le facteur de transcription B entraine la production de la protéine effectrice qui contrôle la voie de l'autre activateur (dans notre exemple A). Ainsi une fois que le réseau a associé les stimuli A et B ensemble (interrupteur en position "on"), l'arrivée, plus tard, du stimulus A (respectivement B) suffit à déclencher la voie B (respectivement A) alors que ce n'était pas le cas avant l'association.
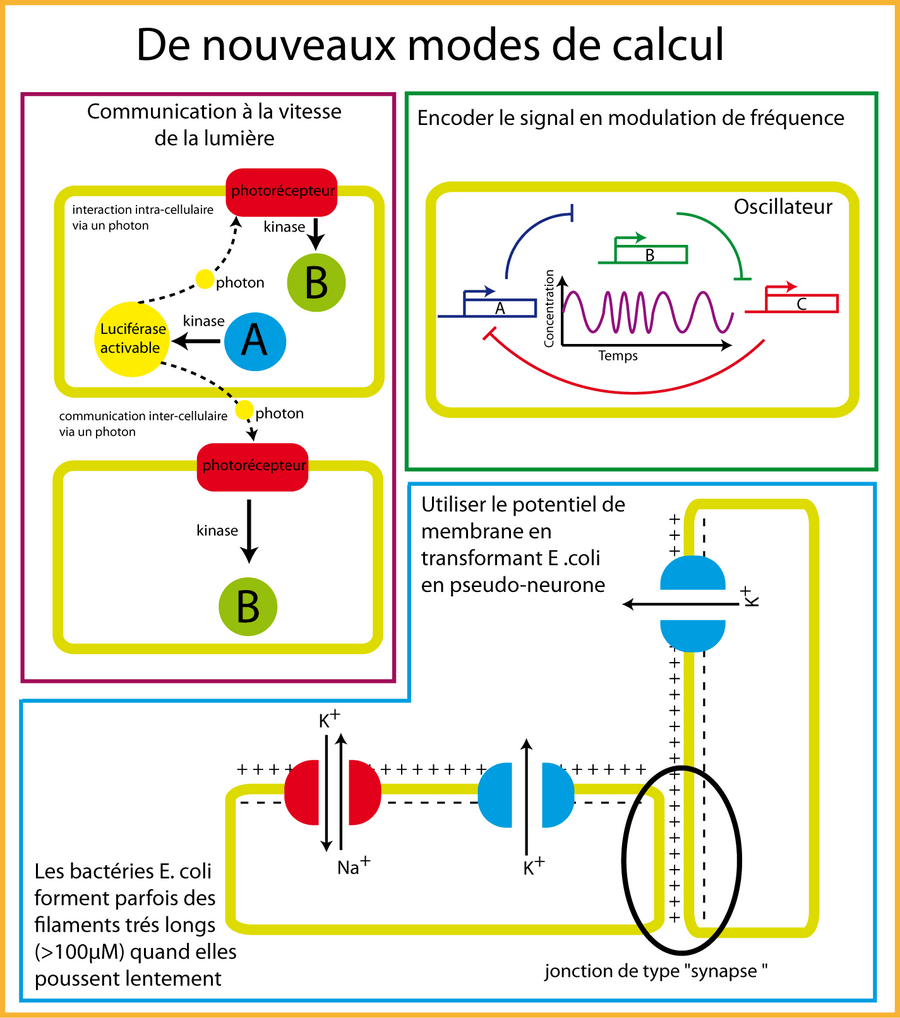
Utiliser d'autres fondements pour le calcul
Les capacités computationnelles des réseaux synthétiques actuels proviennent principalement des réseaux géniques intracellulaires. Nous avons vu qu'il faut élargir ces capacités à la transduction du signal pour gagner en vitesse et aux copies de bactéries clonales pour gagner en parallélisme. Mais on peut encore envisager d'autres formes de calcul :
- Avec la lumière : il existe des enzymes capables d'émettre des photons (la luciferase) et des photorécepteurs (les phytochromes) capables de détecter des photons. Si on réussissait à coupler ces deux systèmes d'émission et de réception (et c'est loin d'être gagné à cause de problèmes quantitatifs), on pourrait peut être réussir à transmettre une information en intra ou intercellulaire via les ondes électromagnétiques (les photons). Autrement dit à faire communiquer deux bactéries à la vitesse de la lumière 😀 .
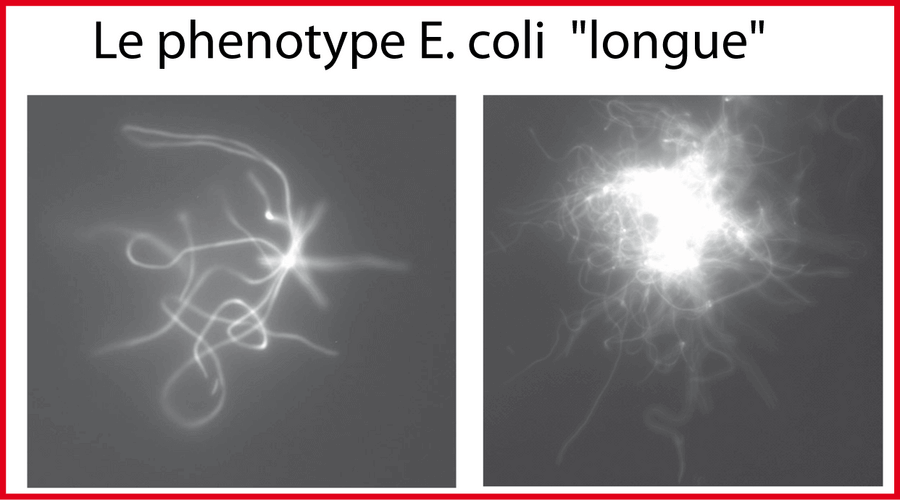
- Avec l'électricité : peut-on transformer E. coli en neurone ? Autrement dit peut-on imaginer utiliser des canaux potassiques, calciques, sodiques pour contrôler l'état de polarisation de la membrane d'E. coli ? puis utiliser cet état de polarisation pour transmettre de l'information entre cellules ? Cette idée de réseau d'E. coli comme réseau de neurones germe plus facilement lorsque l'on observe des bactéries E. coli très longues (>100 μM) (dû sans doute à une croissance très lente à 8--10°C)
- En codant l'information en modulation de fréquence : l'information dans un réseau génique intracellulaire est codée en amplitude. Peut-on faire comme avec les potentiels d'action des neurones qui encodent l'information en modulation de fréquence ? On sait déjà faire des oscillateurs qui génèrent des "spikes". Selon Timothy Lu, cette information de fréquence pourrait être utile pour propager de l'information sur de longues distances car la fréquence est moins susceptible de diminuer au fur et à mesure que la distance s'accroit comparé au nombre absolu de molécules qui forment inexorablement un gradient de concentration.
Les applications à court et moyen terme
Pour des raisons pratiques, je vais découper les applications relatives à la biologie synthétique en deux grandes familles:
- les applications dans le domaine de la santé, du calcul, de l'agriculture, de l'industrie et de l'écologie.
- les applications militaires, les applications relatives à l'augmentation de l'espérance de vie et enfin la question du surhomme.
Le premier point est l'objet de la fin de ce chapitre. Le deuxième point est l'objet des deux chapitres qui suivent.
Dans le premier point, il n'y a pas, il me semble, de questions éthiques, existentielles ou relatives à
l'essence de l'homme. Les bactéries et les "protocellules" n'ont pas de systèmes nerveux. Par
conséquent, leur utilisation n'engendre aucune souffrance. Une bactérie avec un système synthétique
devrait être considérée comme un vulgaire assemblement d'atomes ayant la capacité de
traiter de
l'information. J'ai finalement barré le vulgaire car il me semble que l'on doit tout de même le respect
à nos ancêtres 😀. Ces applications nécessitent cependant quand même l'application du
principe de précaution de manière raisonnable. Notez également qu'il pourrait être néfaste, sur le plan
économique, que ce premier point subisse les conséquences d'un débat sociétal dont l'enjeu serait le
deuxième point. C'est pourquoi je m'attelle à opérer la démarcation. Cette dernière est également
temporelle : le premier point est une question d'années ou de décennies alors que le deuxième point est
une question de demi-siècles ou de siècles. Enfin, il me semble fort probable que la plupart des
chercheurs défendraient âprement le premier point avec pour argument principal la liberté de mener des
recherches pacifistes susceptibles d'augmenter le niveau de confort10 de l'homme. A
l'inverse, la plupart des chercheurs refuseraient de créer des armes biologiques et entrevoient la
question du surhomme et de l'espérance de vie avec le même regard, les mêmes doutes et les mêmes
craintes que le peuple.
Voici les deux principales applications peu polémiques que les biologistes synthétiques entrevoient pour les décennies à venir :
L'usine microbienne
L'industrie chimique fabrique toutes sortes de composés chimiques dont certains sont très chers à synthétiser. L'idée consiste à construire une souche bactérienne qui se charge de la fabrication du dit composé. Le temps de recherche et développement pour construire la souche peut être important car les techniques utilisées (ingénierie du métabolisme, biologie moléculaire et biologie synthétique) sont difficiles. Mais une fois la souche obtenue, on ne risque pas de la perdre : elle est acquise. Il ne reste plus qu'à la placer dans un grand fermenteur pouvant dépasser 1000 mètres cubes avec un milieu de culture contenant divers minéraux ainsi que des sources de carbone et d'azote bon marché. On laisse ensuite les bactéries opérer la transformation (j'imagine que cela est relativement rapide : jours ?) puis on récupère le composé d'intérêt. Les rendements peuvent être conséquents : un pourcentage non négligeable du poids de la source de carbone ajoutée au préalable sera converti en molécule d'intérêt. Si la différence de prix au kilo entre les deux molécules est très importante, le bénéfice le devient aussi. Le composé en question peut être un médicament : par exemple l'artemisinin un médicament anti-malaria (Ro et al. 2006) . Mais ce peut être aussi un colorant (Yan et al. 2005), un simple acide aminé (Park et al. 2007), un bio-fuel (Atsumi and Liao 2008) ou des nano-materiaux (la soie des araignées) (Widmaier et al. 2009). L'ingénierie du métabolisme permet aussi d'envisager de fabriquer des souches chargées de décontaminer un milieu pollué en transformant le composé polluant en un composé non polluant. Pour éviter que ces bactéries, placées dans l'écosystème polluée, ne se propagent au reste de l'environnent de manière non souhaitée, les biologistes développent des technologies de "bio-confinement" pour garder le contrôle de la dissémination de la bactérie. Voir le chapitre sur l'évolution.
La santé
Le développement des systèmes synthétiques de première génération permet d'imaginer développer des thérapeutiques différentes du simple médicament type "molécule chimique active". On peut imaginer fabriquer des souches "bio-senseurs" capables de détecter, par exemple, un problème dans l'intestin (hausse de température, présence d'une bactérie pathogène, d'une toxine, d'un virus) puis d'envoyer un signal (output) détectable (par exemple dans les selles : luminescence, pigments, odeurs) pouvant alerter le patient ou le médecin. Pour cela, il faut déjà accroitre notre connaissance dans un domaine de recherche qui me semble relativement peu exploité: l'écosystème gastro-intestinal. Bien sûr, en plus d'avoir un rôle de senseur passif, les souches bactériennes synthétiques à visée médicale pourraient avoir un rôle plus actif : "attaquer" une souche pathogène (en lui volant tout simplement ses ressources par exemple), dégrader une toxine ou synthétiser des molécules antivirales. Les oscillateurs synthétiques pourraient être utilisés pour permettre la synthèse d'un médicament à intervalle de temps régulier. Dans le cas de pathologies comme le diabète, des boucles de rétroaction pourraient contrôler la production d'insuline par des bactéries en fonction de la concentration extérieure en glucose (mesurée à l'aide des mécanismes de la répression catabolique par exemple) (j'avoue que dans cet exemple, je ne sais pas trop dans quelle partie du corps il faudrait placer la souche).
Enfin, il faut savoir que, après une injection en intraveineuse (ce qui ne pose pas de problème avec une souche non pathogène), certaines souches bactériennes comme E. coli disposent de la faculté de venir se loger automatiquement dans les tumeurs sans que l'on sache exactement pourquoi (Jiang et al., 2010). Ainsi en développant un système synthétique cytotoxique qui détruirait spécifiquement les cellules tumorales, on pourrait imaginer un traitement innovant contre le cancer. Dans sa publication, Jiang injecte des bactéries E. coli en intraveineuse dans des souris. Les bactéries disposent d'un plasmide exprimant la luciferase et un autre permettant l'expression du gène clyA codant pour la cytolysin A, une drogue anti-tumorale. D'une part, les chercheurs identifient les métastases de la souris de manière transcutanée grâce à la luciferase des bactéries venues se loger dans la tumeur. D'autre part, les chercheurs observent une résorption significative des métastases grâce à l'action de la cytolysin A11. Certes il s'agit de résultats très fondamentaux ayant peu de chance de déboucher sur des essais cliniques mais ils ont le mérite de pointer du doigt, au biologiste synthétique, une direction potentiellement prometteuse.
Bibliographie
- Atsumi, S. and J. C. Liao (2008). "Metabolic engineering for advanced biofuels production from Escherichia coli." Curr Opin Biotechnol 19(5): 414-9.
- Balagadde, F. K., H. Song, et al. (2008). "A synthetic Escherichia coli predator-prey ecosystem." Mol Syst Biol 4: 187.
- Basu, S., Y. Gerchman, et al. (2005). "A synthetic multicellular system for programmed pattern formation." Nature 434(7037): 1130-4.
- Chang, D. E., S. Leung, et al. (2009). "Building biological memory by linking positive feedback loops." Proc Natl Acad Sci U S A.
- Danino, T., O. Mondragon-Palomino, et al. (2010). "A synchronized quorum of genetic clocks." Nature 463(7279): 326-30.
- Datsenko, K. A. and B. L. Wanner (2000). "One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products." Proc Natl Acad Sci U S A 97(12): 6640-5.
- Del Vecchio, D., A. J. Ninfa, et al. (2008). "Modular cell biology: retroactivity and insulation." Mol Syst Biol 4: 161.
- Elf, J., G. W. Li, et al. (2007). "Probing transcription factor dynamics at the single-molecule level in a living cell." Science 316(5828): 1191-4.
- Elowitz, M. B. and S. Leibler (2000). "A synthetic oscillatory network of transcriptional regulators." Nature 403(6767): 335-8.
- Friedland, A. E., T. K. Lu, et al. (2009). "Synthetic gene networks that count." Science 324(5931): 1199-202.
- Gardner, T. S., C. R. Cantor, et al. (2000). "Construction of a genetic toggle switch in Escherichia coli." Nature 403(6767): 339-42.
- Gibson, D. G., J. I. Glass, et al. "Creation of a bacterial cell controlled by a chemically synthesized genome." Science 329(5987): 52-6.
- Glass, J. I., N. Assad-Garcia, et al. (2006). "Essential genes of a minimal bacterium." Proc Natl Acad Sci U S A 103(2): 425-30.
- Hashimoto, M., T. Ichimura, et al. (2005). "Cell size and nucleoid organization of engineered Escherichia coli cells with a reduced genome." Mol Microbiol 55(1): 137-49.
- Jiang, S. N., T. X. Phan, et al. "Inhibition of Tumor Growth and Metastasis by a Combination of Escherichia coli-mediated Cytolytic Therapy and Radiotherapy." Mol Ther.
- Levskaya, A., A. A. Chevalier, et al. (2005). "Synthetic biology: engineering Escherichia coli to see light." Nature 438(7067): 441-2.
- Li, G. W. and J. Elf (2009). "Single molecule approaches to transcription factor kinetics in living cells." FEBS Lett 583(24): 3979-83.
- Lu, T. K., A. S. Khalil, et al. (2009). "Next-generation synthetic gene networks." Nat Biotechnol 27(12): 1139-50.
- Noireaux, V. and A. Libchaber (2004). "A vesicle bioreactor as a step toward an artificial cell assembly." Proc Natl Acad Sci U S A 101(51): 17669-74.
- Park, J. H., K. H. Lee, et al. (2007). "Metabolic engineering of Escherichia coli for the production of L-valine based on transcriptome analysis and in silico gene knockout simulation." Proc Natl Acad Sci U S A 104(19): 7797-802.
- Posfai, G., G. Plunkett, 3rd, et al. (2006). "Emergent properties of reduced-genome Escherichia coli." Science 312(5776): 1044-6.
- Ro, D. K., E. M. Paradise, et al. (2006). "Production of the antimalarial drug precursor artemisinic acid in engineered yeast." Nature 440(7086): 940-3.
- Sauro, H. M. (2008). "Modularity defined." Mol Syst Biol 4: 166.
- Sohka, T., R. A. Heins, et al. (2009). "An externally tunable bacterial passe-bande filter." Proc Natl Acad Sci U S A 106(25): 10135-40.
- Stricker, J., S. Cookson, et al. (2008). "A fast, robust and tunable synthetic gene oscillator." Nature 456(7221): 516-9.
- Tabor, J. J., H. M. Salis, et al. (2009). "A synthetic genetic edge detection program." Cell 137(7): 1272-81.
- Widmaier, D. M., D. Tullman-Ercek, et al. (2009). "Engineering the Salmonella type III secretion system to export spider silk monomers." Mol Syst Biol 5: 309.
- Yan, Y., J. Chemler, et al. (2005). "Metabolic engineering of anthocyanin biosynthesis in Escherichia coli." Appl Environ Microbiol 71(7): 3617-23.
- Yu, D., H. M. Ellis, et al. (2000). "An efficient recombination system for chromosome engineering in Escherichia coli." Proc Natl Acad Sci U S A 97(11): 5978-83.
Notes de bas de page
- Source Wikipedia : article sur la biologie synthétique.
- Source Rapport de l'Académie Royale d'Ingénierie de l'UK sur la biologie synthétique p. 33. L'emphase : c'est moi qui souligne.
- Un an plus tard, j'ai un doute sur cette manière de présenter les choses}
- Bien sûr, c'est les étudiants qui déterminent leur sujet
- Mais pas seulement bien sûr et beaucoup de thésards, post-docs, chercheurs font aussi de la biologie synthétique.
- Thomas Kuhn, La structure des révolutions scientifiques, voir le chapitre d'épistémologie.
- Excusez-moi si cela peut sembler irrévérencieux. Ce n'est pas mon intention : seul le concept Kuhnien m'intéresse ici.
- Notez que je n'ai jamais participé à une compétition IGEM : mon avis est donc relativement impartial. Cependant, il y a une équipe IGEM dans mon laboratoire et j'ai pu constater la grande motivation de ses membres.
- Mais il existe d'autres méthodes possibles pour contrôler le déplacement d'une bactérie (voir le paragraphe sur le chimiotactisme dans le chapitre sur E. coli).
- Ce mot "confort" peut choquer quand il s'agit de santé. Cependant, je l'utilise ici car la notion de santé est trop proche/difficilement dissociable des notions plus polémiques d'allongement de l'espérance de vie et donc de la disparition de la mort. Confort intègre donc ici une idée sur laquelle nous sommes, il me semble, tous d'accord : nous ne souhaitons pas "souffrir" physiquement avant de mourir.
- Le traitement est effectué en combinaison avec de la radiothérapie ce qui rend la publication moins " sexy" mais ne discrédite en rien les résultats.