La bactérie Escherichia coli
Préambule
Ce chapitre est relativement facile d'accès. Il est écrit pour les non-biologistes (par exemple les mathématiciens) qui veulent se forger rapidement une image globale d'une bactérie comme E. coli. En une quinzaine de pages, le lecteur se fera une idée des grands domaines d'étude sur E. coli autres que les réseaux de régulation (réservé au chapitre suivant). Je l'ai aussi écrit en pensant aux jeunes biologistes synthétiques qui pourraient potentiellement y puiser quelques idées intéressantes: par exemple, je leur rappelle qu'E. coli peut se développer dans des cellules eucaryotes et détourner le cytosquelette d'actine pour se déplacer.
Différence entre procaryotes et eucaryotes
Pour éviter toutes confusions dans l'esprit d'un lecteur peu familier avec la biologie, je souhaite rappeler ici quelques différences essentielles entre cellules procaryotes (grossièrement les bactéries dont E. coli) et les cellules eucaryotes (par exemple, les cellules humaines ou les levures). Première différence: la taille. E. coli mesure 2µm de long sur 0.5µm de large (1µm3), c'est-à-dire en général 10 à 100 fois plus petite qu'une cellule eucaryote. Les bactéries n'ont pas de noyau et en général elles ne possèdent qu'un seul chromosome circulaire. L'absence de noyau a une conséquence évidente: la transcription se fait directement dans le cytoplasme. De plus, Il n'y a pas d'intron donc pas d'épissage. Notons aussi l'absence de structures cytoplasmiques comme le réticulum endoplasmique, les mitochondries, les chloroplastes ou le cytosquelette (microtubule). Enfin, le mode de reproduction d'E. coli est la division simple alors que les cellules eucaryotes se divisent par mitose (multiplication conforme de la cellule) ou par méiose (formation de gamètes). Ainsi E. coli est un organisme très simple comparé aux eucaryotes. L'absence de compartiments intracellulaires est probablement la différence primordiale. En tout cas du point de vue du modélisateur.
Réplication, transcription, traduction et repliement des protéines
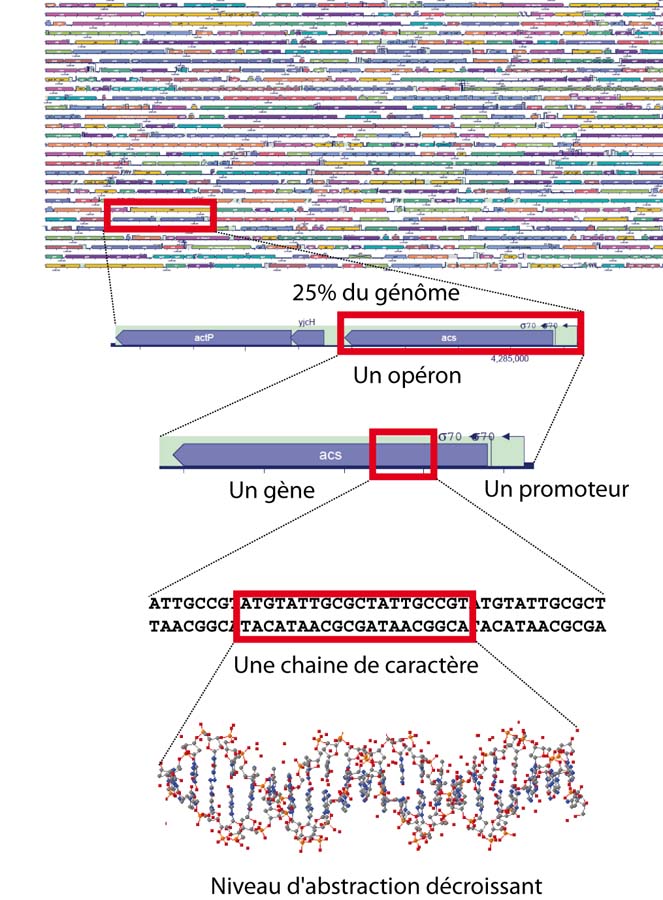
E. coli possède un chromosome circulaire de 4.6 millions de paire de bases (pb). Environ 4500 gènes (soit en moyenne 1000 pb par gène) ont été localisés et 85% ont une fonction connue. Restent environ 850 dont la fonction est inconnue (Merlin et al. 2002). Plus ce chiffre diminuera, plus la difficulté pour déterminer leurs fonctions augmentera mais plus on risquera de découvrir de nouveaux paradigmes dans le fonctionnement d'une cellule. On estime que E. coli dispose d'environ 303 gènes essentiels c'est-à-dire dont la disruption/délétion entraîne la non viabilité de la cellule (Baba et al. 2006).
Le chromosome se trouve sous forme super-enroulé dans la bactérie. Le super enroulement de l'ADN est finement contrôlé par des enzymes: les topoisomerases cibles de plusieurs antibiotiques dont les fluoroquinolones (Nollmann et al. 2007). Des protéines ressemblant à des histones (HU, HNS, fis, IHF) structurent également le chromosome mais les procaryotes ne possèdent pas d'histone à proprement parler (Ali Azam et al. 1999).
La réplication du chromosome d'E. coli dure approximativement 42 minutes et débute au niveau d'une origine de réplication unique. La vitesse de réplication de l'ADN polymérase est de 1000pb.s-1. Parfois, cette dernière fait une erreur et introduit une mutation à une fréquence de 10-7 mais des mécanismes de correction des erreurs interviennent pour diminuer cette fréquence. Au final, E. coli fait des erreurs à une fréquence de l'ordre de 10-9 par nucléotide répliqué. Notez que si cette fréquence était nulle, E. coli ne pourrait pas évoluer et ne pourrait donc pas survivre. En effet, bien que ces mutations sont la plupart du temps soit silencieuses soit délétères, dans de rares cas, elles sont bénéfiques ce qui leurs permet d'être sélectionnées au cours de l'évolution et donc d'être fixées. Ces mutations peuvent apparaître dans un gène pour modifier la fonction de la protéine correspondante mais aussi en dehors des gènes, par exemple dans les zones promotrices pour modifier le réseau de régulation. Un informaticien ou mathématicien peut se représenter ces mécanismes évolutifs comme une heuristique de recherche d'optimum global. Un paramètre varie aléatoirement (les mutations) et éloigne le plus souvent de la solution optimale. Cependant, parfois la variation permet de s'échapper d'un optimum local. Dans ce problème d'optimisation, la fonction optimisée par la nature est le fitness de la bactérie défini comme la capacité d'un individu à se reproduire. Nous reviendrons largement, dans le chapitre sur l'évolution, sur le rôle et l'utilisation de l'évolution pour "tuner" un réseau de régulation génique .
Le chromosome contient de nombreux opérons qui sont des groupements de gènes transcrits ensemble. L'ARN polymérase, qui transcrit les gènes en ARN, se fixe au niveau du promoteur: une région de l'ordre de 100pb située à proximité d'un gène et indispensable à la transcription. La fixation et l'activation de l'ARN polymérase sont contrôlées par des facteurs de transcription qui se fixent sur leur site de liaison spécifique (10pb) au niveau de la région promotrice. Les facteurs de transcription peuvent être soit activateurs, soit répresseurs. Plus de 196 facteurs de transcription sont décrits parmi les 4500 gènes que compte E. coli K-12 (Keseler et al. 2009). Certains facteurs de transcription dit "régulateurs globaux" sont capables de contrôler la transcription de plusieurs dizaines voire centaines de gènes (par exemple Crp, ArcA, FruR, Fnr, Mlc)(Perrenoud and Sauer 2005). La transcription d'un gène prend environ 1 minute. Elle est ciblée par plusieurs antibiotiques dont la rifampicine.
Puis l'ARNm est traduit par un ribosome (environ 104 par cellules) en environ 2 minutes. Le ribosome est la cible de nombreux antibiotiques comme le chloramphénicol. Notez qu'un ARNm peut être traduit plusieurs fois mais sa durée de vie est très faible, de l'ordre de 2 à 5 minutes. L'arrêt de la transcription d'un gène peut donc impacter assez rapidement la concentration de la protéine résultante. E. coli possède environ 4 millions de protéines (taille moyenne 5nm) par cellule. Chaque type de protéine se retrouve en moyenne à la concentration de 1nM c'est-à-dire quelques unités par cellules ; cependant certaines sont présentes en centaines de milliers de copies dans une cellule.
Une fois traduit, un polypeptide se replie dans sa structure tridimensionnelle caractéristique dans laquelle il est fonctionnel. Le mécanisme du repliement des protéines n'est pas encore compris cependant la conformation finale dite "native" est probablement énergétiquement favorable. Les repliements rapides prennent quelques microsecondes mais d'autres demandent plusieurs minutes à plusieurs heures pour se produire. Certaines protéines sont assistées dans leurs repliements par d'autres protéines dites chaperonnes (GroEL/GroEs; DnaK/DnaJ) qui évitent ainsi les phénomènes d'agrégation de protéines mal repliées (Ben-Zvi and Goloubinoff 2001). Enfin, il existe aussi un ensemble de protéines (ClpX,clpA) capables de reconnaître et de déplier des protéines marquées ou incorrectement repliées pour leur laisser une seconde chance de repliement ou au contraire les entrainer vers la dégradation (protéase clpP, lon) (Farrell et al. 2005).
Les frontières de la vie: les membranes
La frontière entre le milieu extérieur et E. coli ne fait que quelques centaines d'atomes d'épaisseur. Mais cette frontière est loin d'être simple. E. coli est une bactérie dite à gram négatif dont la structure s'organise en trois grandes parties, soit, de l'intérieur vers l'extérieur, la membrane plasmique (bicouche lipidique), l'espace périplasmique et la membrane externe (bicouche lipidique). Les deux derniers forment ce qu'on appelle la paroi dont la formation est la cible de plusieurs antibiotiques comme la pénicilline. Cette frontière assure plusieurs fonctions. Elle assure le maintien de la forme de la bactérie et le maintien d'un environnement osmotiquement stable. Elle empêche les molécules du milieu extérieur de rentrer et garde les 60 millions de molécules intracellulaires proches les unes des autres pour permettre aux réactions de se dérouler rapidement.
Cependant, la bactérie a besoin d'une connexion avec le monde extérieur. Pour survivre et croître, un organisme doit importer du matériel brut et se débarrasser des déchets produits. E. coli dispose de dizaines de milliers de transporteurs protéiques enchâssés dans la membrane externe et dans la membrane plasmique. Ces pores, canaux ou pompes transportent, suivant le cas, dans un sens ou dans l'autre et de manière active ou passive. Le plus souvent, ces transporteurs sont spécifiques d'une molécule donnée. La discrimination se fait le plus souvent en fonction de leur forme. Plus de 254 transporteurs sont décrits parmi les 4500 gènes que compte E. coli K-12 (Keseler, Bonavides-Martinez et al. 2009). Dans la membrane, on trouve également des senseurs protéiques. Ces derniers détectent un changement dans l'environnement extérieur (par exemple, la présence d'une molécule) et transmettent l'information (le signal) à l'intérieur de la cellule le plus souvent par le biais d'une cascade de modifications chimiques (par exemple des phosphorylations) aboutissant à une modification de l'expression génique.
Le métabolisme
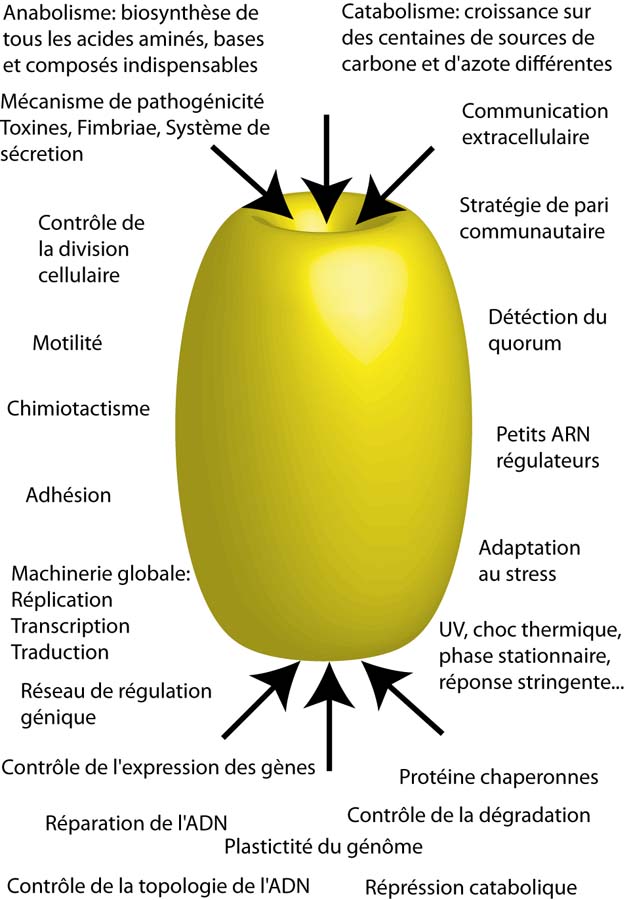
La présence d'une frontière, d'un chromosome et d'une machinerie de réplication, transcription et traduction ne suffit pas pour faire "émerger" la vie. La cellule représente un état hautement organisé de la matière: maintenir cet ordre tout en étant soumis aux principes de la thermodynamique nécessite la mise en place de structures permettant d'utiliser l'énergie (on crée de l'ordre au niveau de la cellule mais, globalement, l'entropie augmente). La cellule est donc un système thermodynamiquement ouvert nécessitant un métabolisme c'est-à-dire un ensemble de réactions chimiques qui se déroulent de manière ininterrompue. Le métabolisme est usuellement divisé en deux catégories: les processus de dégradation (catabolisme) par exemple pour casser des molécules organiques et produire de l'énergie. Et des processus de synthèse organique (anabolisme) pour biosynthétiser des composés absents dans le milieu de culture comme des acides aminés ou des bases.
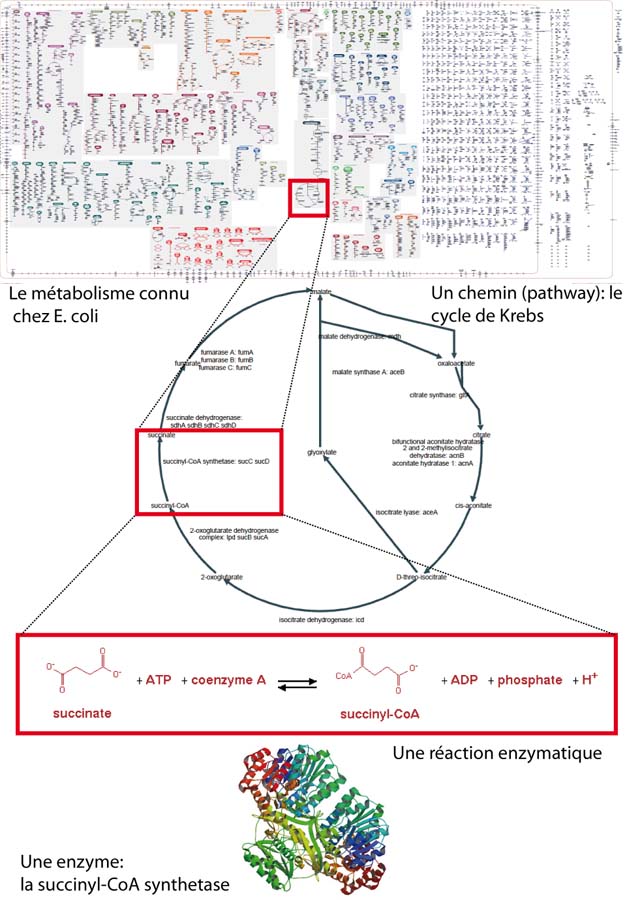
Les réactions chimiques sont organisées en chemins (pathway) métabolique dans lesquels un métabolite est transformé en un autre métabolite grâce à une série de réactions chimiques catalysées par une série d'enzymes. Certains chemins métaboliques sont très conservés à travers l'évolution. Tout comme le code génétique ou le dogme ADN--ARN--protéine, la glycolyse et le cycle de Krebs sont présents aussi bien chez E. coli que chez l'éléphant. La première espèce dont le métabolisme a été décrit en détail est justement E. coli. Cela a été un travail long et fastidieux qui a pris des décennies. Aujourd'hui, on compte plus de 1397 gènes codant pour des enzymes chez cet organisme (Keseler, Bonavides-Martinez et al. 2009).
La division cellulaire
La bactérie E. coli se divise par fission binaire. Dans un milieu riche, à 37°C sous agitation (respiration aérobie), E. coli se divise toutes les 20 minutes. Sa croissance est exponentielle. Autrement dit, si la croissance ne s'arrêtait pas, faute de nutriments ou suite à l'accumulation de déchets, E. coli recouvrirais la terre en quelques jours.
Le processus de division inclut la réplication du chromosome, la ségrégation des deux chromosomes de part et d'autre de chaque pôle de la bactérie et la formation d'un septum permettant la séparation en deux cellules filles. Cette séparation débute avec l'assemblage d'un anneau septal composé d'une douzaine de protéines. Le composé majeur de cet anneau septal est la protéine FtsZ, l'homologue procaryote de la tubuline, une des plus importantes protéines du cytosquelette chez les eucaryotes. La polymérisation de FtsZ forme un anneau contractile qui scinde la bactérie en deux cellules filles. Une souche, sans le gène ftsZ (un mutant de délétion), est en effet incapable de se diviser et forme de longs filaments pouvant faire plusieurs dizaines de micromètres. Une régulation spatiale et temporelle très fine assure le bon déroulement du processus de division. En effet, il faut inhiber la formation de l'anneau septal tant que la bactérie est trop petite et s'assurer que l'assemblage de cet anneau se crée bien au milieu de la bactérie et non au pôle (Margolin 2001).
L'adaptation au stress
Une cellule bactérienne n'a que très peu de possibilités pour choisir et modifier son environnement. Il lui faut donc disposer de mécanismes qui lui permettent de maintenir son homéostasie en répondant rapidement et efficacement à une variété de stress environnementaux qui pourraient menacer son intégrité. Pour cela, la bactérie dispose de senseurs et d'effecteurs capables de faire face, par exemple, à un changement brutal de température, à une forte irradiation ou à l'épuisement des nutriments dans le milieu. En général, ces mécanismes ont un impact négatif sur le taux de croissance. Ainsi, en fonction de conditions extérieures, la bactérie module nécessité de reproduction et nécessité de protection.
Réponse aux chocs thermiques
Un choc thermique a pour conséquence l'accumulation de protéines mal repliées. Ces dernières exposent des résidus hydrophobes à leur surface ce qui entraine leurs agrégations. Cette agrégation menace sérieusement l'organisation et le fonctionnement des composants cytoplasmiques. L'ARN polymérase d'E. coli est composée d'un coeur enzymatique (5 sous-unités) responsable de la polymérisation de l'ARN et d'une sixième sous-unité appelée facteur sigma qui permet la reconnaissance des régions promotrices et l'initiation de la transcription. 7 facteurs sigma différents ont été identifiés chez E. coli et chacun d'eux régule la transcription d'un groupe spécifique de gènes. En cas de choc thermique, la concentration du facteur sigma dit "32" augmente très fortement. Ce facteur sigma se lie au coeur de l'ARN polymérase et permet la transcription de 2 types de gènes: des chaperonnes qui assistent le repliement des protéines et de protéases qui dégradent les protéines mal repliées (Arsene et al. 2000). La mise en place de cette régulation permet donc de limiter les effets négatifs du choc thermique (l'agrégation des protéines).
Réponse à une carence en acides aminés
La réponse stringente est le nom donné à la réponse au stress qui apparait lors d'une carence en acides aminés. Lorsque celle-ci survient, une molécule le (p)ppGpp est produite en grande quantité. Cette molécule inhibe, en partie, la transcription et décroit ainsi la synthèse de la machinerie de traduction (ARNt, ARNr) dont la fonction (la synthèse de protéines) est inutile sans acide aminés (les briques de base des protéines). Cet "alarmone" (le (p)ppGpp) réoriente ainsi l'utilisation des ressources énergétiques vers d'autres processus prioritaires comme la bio-synthèse des acides aminés (Chatterji and Ojha 2001).
Réponse à une exposition UV
L'exposition de E. coli à la lumière ultra-violette endommage fortement l'ADN ce qui conduit à l'accumulation de simple brin d'ADN. Pour répondre à ce stress, la bactérie possède un système dit SOS, découvert par Miroslav Radman, qui permet à la bactérie de réparer, en partie, son ADN (Radman 1974). Lors d'une exposition aux UV, la protéine RecA se fixe aux simples brins d'ADN apparus et induit le clivage d'une autre enzyme nommé LexA. La disparition de LexA induit la transcription d'une quarantaine de gènes dont la plupart sont impliqués dans la protection, la réparation et la réplication de l'ADN (Janion 2008).
Motilité, agrégation, biofilm et quorum sensing
La bactérie E. coli n'est pas condamnée à rester immobile. Pour assurer sa mobilité, E. coli dispose d'une dizaine de flagelles qui tournent comme une hélice de bateau ce qui assure la propulsion. Un flagelle est une structure cylindrique creuse et semi-rigide composé d'une protéine, la flagelline, formant un filament d'une dizaine de micromètres de longueur pour 20nm d'épaisseur. Le filament est ancré dans la membrane plasmique au niveau d'une embase où se trouve le moteur moléculaire. Ce dernier tire son énergie du flux de protons qui traversent la membrane dû au gradient de concentration existant de part et d'autre. Ce moteur assure la rotation du flagelle à plus de 1000 révolutions par seconde ce qui permet à la bactérie d'atteindre une vitesse allant jusqu'à 30µm/s. La motilité des bactéries est, par exemple, très utile pour se déplacer en direction d'une source de nutriments ou au contraire pour s'éloigner d'une substance toxique. Nous reviendrons en détail sur ce phénomène appelé chimiotactisme.
La bactérie E. coli n'est pas condamnée à subir les flux de liquides. En effet, les bactéries E. coli sont capables d'adhérer à la plupart des surfaces (PVC, polycarbonate, verre...) et de s'agréger entre elles. Pour adhérer, les bactéries utilisent des appendices protéiques plus petits et plus fins que les flagelles: les fimbriae présents en plusieurs centaines d'exemplaires sur chaque cellule. On utilise le terme "biofilm" lorsque on parle d'une communauté de micro-organismes adhérant entre eux et à une surface, et marquée par la sécrétion d'une matrice adhésive et protectrice. Les micro-organismes présents dans la communauté sont souvent capables d'agir de façon coordonnée. Il existe ainsi des mécanismes régulateurs chargés de synchroniser l'expression de gènes particuliers au sein d'une population. Ce mécanisme dit de "quorum sensing" utilise la capacité des bactéries à communiquer avec leurs congénères via des signaux moléculaires. E. coli produit une molécule le 4,5-dihydroxy-2,3-pentanedione dit "auto-inducteur". Lorsque la densité de population est faible, la diffusion réduit rapidement la concentration de l'auto-inducteur dans le milieu. A l'inverse, lorsque cette densité augmente, par exemple dans un milieu confiné comme un biofilm, la concentration de l'auto-inducteur dépasse un seuil critique qui est alors perçu par chaque bactérie en même temps. Cela permet de synchroniser l'expression génique pour l'ensemble de la population qui se comporte alors un peu à la manière d'un organisme pluricellulaire et non plus comme une simple somme d'individus indépendants.
Une des publications présente dans cette thèse décrit le fonctionnement d'un système synthétique de communication intercellulaire basé sur une petite molécule: l'AMPc.
La flore intestinale
La flore intestinale est l'écosystème principal de la bactérie E. coli. Elle contient 1014 bactéries c'est-à-dire dix fois plus que le nombre de cellules humaines dans le corps. On pense qu'il y a environ 1000 espèces qui peuplent nos intestins. E. coli représenterait moins de 1 bactérie sur 100: en effet plus de 99% de nos bactéries seraient des bactéries anaérobies strictes (Firmicutes et les Bacteroidetes). Il est ainsi très difficile d'étudier cet écosystème car la majorité des espèces bactériennes ne peuvent pas être cultivées en présence d'oxygène. Reproduire l'écosystème d'un colon n'est donc pas chose aisée. Et il est encore plus difficile d'étudier les relations qu'entretiennent les différentes espèces entre elles ou avec leur hôte. Ces associations peuvent être répertoriées en commensalisme, mutualisme ou parasitisme. Nous décrivons ci-dessous chacune d'elles.
Le commensalisme est un type d'interaction entre deux êtres vivants dans laquelle l'hôte fournit une partie de sa propre nourriture au commensal sans obtenir de contrepartie évidente de ce dernier. On pense qu'un certains nombre d'espèces bactériennes peuplant nos intestins entretiennent ce type de relation dont le bénéfice n'est pas réciproque.
Cependant, il existe aussi des interactions entre deux ou plusieurs espèces, dans laquelle le symbiote et l'hôte tirent tous les deux profit de cette relation. On qualifie ce type d'association de mutualisme. Elle expliquerait la cohabitation de plus de 1000 espèces différentes dans nos intestins. Selon un certain paradigme, on peut envisager ces différentes espèces comme les maillons d'un métabolisme extracellulaire (comme les enzymes sont les maillons du métabolisme intracellulaire)(Lehours 2010). Cette flore intestinale, composée de plus de 3.3 millions de gènes (Qin et al.) (150 fois plus que le génome humain) serait, selon certains chercheurs, un organe "oublié" qui aurait de nombreuses fonctions utiles pour les humains. En effet, bien qu'on puisse vivre sans flore intestinale (la gnotobiologie), les microorganismes accomplissent de nombreuses fonctions utiles comme:
- la fermentation de résidus non digestes
- la production de vitamines (E. coli produit la vitamine k2)
- le contrôle de la prolifération des cellules épithéliales et leur différentiation
- le développement et l'entrainement du système immunitaire
- la protection contre les pathogènes (l'effet barrière)
On sait également que la composition et l'abondance relative des espèces bactériennes qui colonisent l'intestin varient d'un individu à l'autre. En effet, une étude a révélé que la flore intestinale de personnes obèses aurait une capacité accrue à extraire l'énergie de l'alimentation (Turnbaugh et al. 2006).
Le parasitisme est une relation biologique où un des protagonistes, le parasite, tire profit (en se nourrissant, en s'abritant ou en se reproduisant) d'une espèce tiers, l'hôte, aux dépens de celui-ci. Certaines souches pathogènes d'E. coli sont responsables de gastro-entérites, d'infections urinaires, de méningites et de septicémies. La recherche d'un vaccin continue (Moriel et al.). Je décris dans le paragraphe qui suit les mécanismes de virulence de 4 types de souches.
Les souches d'E. coli pathogènes
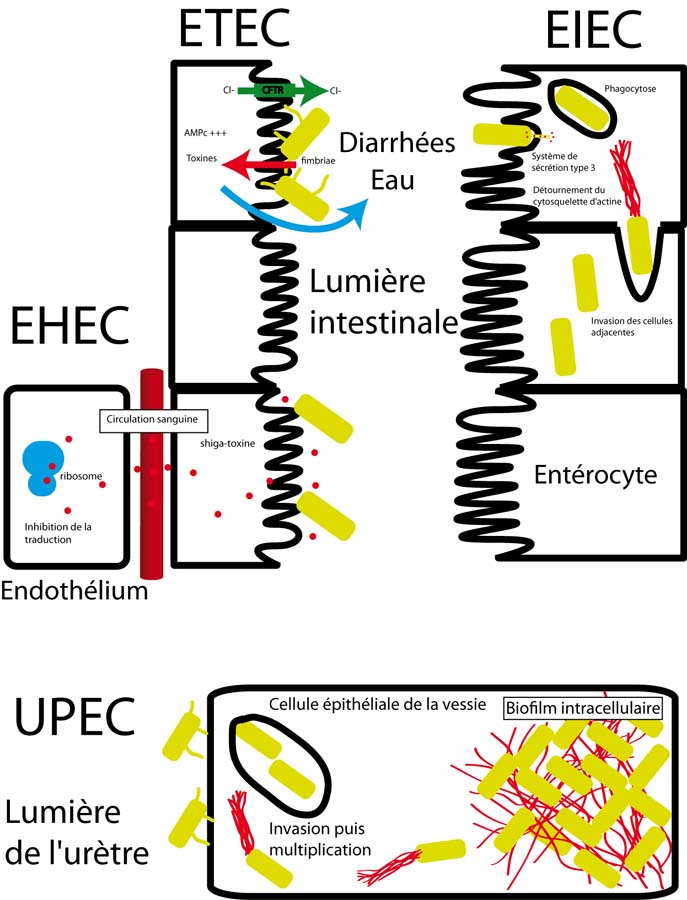
Les E. coli entérotoxigéniques (ETEC) sont responsables de plus de 380 000 morts/an (source O.M.S). Les décès touchent principalement des enfants de moins de 3 ans qui meurent de déshydratation suite à des diarrhées très sévères (Steffen et al. 2005). Ces décès ne sont pas dus à l'absence de traitements existants mais plutôt à l'absence de structures médicales en mesure de soigner les jeunes patients dans les pays touchés. Le pouvoir pathogène des ETEC est très similaire à celui du cholera. Il s'explique essentiellement par la présence de fimbriaes (qui permettent l'adhésion aux cellules intestinales) et la sécrétion des toxines. Une des toxines, après endocytose, entraine une forte augmentation de la concentration d'une molécule (l'AMPc) ce qui conduit à la phosphorylation d'un transporteur membranaire nommé CFTR. Cette action se traduit par une sécrétion d'ions chlorure ce qui provoque la diffusion osmotique d'eau vers la lumière intestinale puis la diarrhée et potentiellement la mort.
Les E. coli entéroinvasives (EIEC) sont responsables de diarrhées aqueuses qui évoluent rapidement en dysenterie (selles contenant du sang et du mucus). On pense que les mécanismes d'invasion des EIEC sont très similaires à ceux des bactéries Shigella. Elles expriment au niveau de leur membrane, un système de sécrétion dit de type 3 qui agit comme une seringue et injecte des protéines dites " invasines". Ces dernières permettront l'invasion puis la multiplication des bactéries dans le cytoplasme des cellules épithéliales du colon. Les bactéries détournent ensuite la polymérisation de l'actine pour pouvoir se propulser et se disséminer de cellules en cellules. L'invasion puis la destruction de la muqueuse intestinale déclenchent une intense réaction inflammatoire.
Les E. coli entérohémorragiques (EHEC) sont responsables de colites hémorragiques et parfois de défaillance rénale. Des épidémies à EHEC se sont déclarées suite à l'ingestion de viandes contaminées et insuffisamment cuites (hamburger). Les EHEC produisent une toxine (la Shiga-toxine) qui peut être transférée dans la circulation systémique puis qui peut inactiver la sous unité 60S des ribosomes ce qui bloque la traduction et entraine la mort des cellules de l'endothélium. Une des conséquences possibles est le syndrome hémolytique et urémique (SHU) pouvant conduire à la mort.
Les E. coli uropathogènes (UPEC) sont responsables de 90% des infections urinaires. Ces bactéries utilisent les fimbriaes pour se lier aux cellules endothéliales et coloniser ensuite la vessie. Les hémolysines produites entrainent la lyse des cellules des voies urinaires. Les UPEC forment des communautés bactériennes intracellulaires dans les cellules de l'urothélium ce qui leur permet d'échapper au système immunitaire. Elles peuvent également former des biofilms intra et extracellulaires ce qui leur confère une meilleure résistance aux antibiotiques. Ces deux phénomènes expliqueraient la chronicité souvent observée dans les infections des voies urinaires (Blango and Mulvey; Cegelski et al. 2008).
Bibliographie
- Ali Azam, T., A. Iwata, et al. (1999). "Growth phase-dependent variation in protein composition of the Escherichia coli nucleoid." J Bacteriol 181(20): 6361-70.
- Alon, U. (2006). An introduction to system biology, Chapman and Hall/CRC.
- Arsene, F., T. Tomoyasu, et al. (2000). "The heat shock response of Escherichia coli." Int J Food Microbiol 55(1-3): 3-9.
- Baba, T., T. Ara, et al. (2006). "Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection." Mol Syst Biol 2: 2006 0008.
- Ben-Zvi, A. P. and P. Goloubinoff (2001). "Review: mechanisms of disaggregation and refolding of stable protein aggregates by molecular chaperones." J Struct Biol 135(2): 84-93.
- Blango, M. G. and M. A. Mulvey "Persistence of uropathogenic Escherichia coli in the face of multiple antibiotics." Antimicrob Agents Chemother 54(5): 1855-63.
- Cegelski, L., G. R. Marshall, et al. (2008). "The biology and future prospects of antivirulence therapies." Nat Rev Microbiol 6(1): 17-27.
- Chatterji, D. and A. K. Ojha (2001). "Revisiting the stringent response, ppGpp and starvation signaling." Curr Opin Microbiol 4(2): 160-5.
- Farrell, C. M., A. D. Grossman, et al. (2005). "Cytoplasmic degradation of ssrA-tagged proteins." Mol Microbiol 57(6): 1750-61.
- Janion, C. (2008). "Inducible SOS response system of DNA repair and mutagenesis in Escherichia coli." Int J Biol Sci 4(6): 338-44.
- Keseler, I. M., C. Bonavides-Martinez, et al. (2009). "EcoCyc: a comprehensive view of Escherichia coli biology." Nucleic Acids Res 37(Database issue): D464-70.
- Lehours, A.-C. (2010). "les bactéries du lac pavin " Pour la science.
- Margolin, W. (2001). "Spatial regulation of cytokinesis in bacteria." Curr Opin Microbiol 4(6): 647-52.
- Merlin, C., S. McAteer, et al. (2002). "Tools for characterization of Escherichia coli genes of unknown function." J Bacteriol 184(16): 4573-81.
- Moriel, D. G., I. Bertoldi, et al. "Identification of protective and broadly conserved vaccine antigens from the genome of extraintestinal pathogenic Escherichia coli." Proc Natl Acad Sci U S A.
- Nollmann, M., N. J. Crisona, et al. (2007). "Thirty years of Escherichia coli DNA gyrase: from in vivo function to single-molecule mechanism." Biochimie 89(4): 490-9.
- Perrenoud, A. and U. Sauer (2005). "Impact of global transcriptional regulation by ArcA, ArcB, Cra, Crp, Cya, Fnr, and Mlc on glucose catabolism in Escherichia coli." J Bacteriol 187(9): 3171-9.
- Qin, J., R. Li, et al. "A human gut microbial gene catalogue established by metagenomic sequencing." Nature 464(7285): 59-65.
- Radman, M. (1974). "Phenomenology of an inducible mutagenic DNA repair pathway in Escherichia coli: SOS repair hypothesis." Molecular and Environmental aspects of mutagenesis.
- Steffen, R., F. Castelli, et al. (2005). "Vaccination against enterotoxigenic Escherichia coli, a cause of travelers' diarrhea." J Travel Med 12(2): 102-7.
- Sundararaj, S., A. Guo, et al. (2004). "The CyberCell Database (CCDB): a comprehensive, self-updating, relational database to coordinate and facilitate in silico modeling of Escherichia coli." Nucleic Acids Res 32(Database issue): D293-5.
- Turnbaugh, P. J., R. E. Ley, et al. (2006). "An obesity-associated gut microbiome with increased capacity for energy harvest." Nature 444(7122): 1027-31.