Evolution (I)
Préambule
Pourquoi consacrer un chapitre entier à l'évolution dans une thèse qui traite des réseaux de régulation ? Je vais répondre à cette question par une autre question.
"Pourquoi le réseau de régulation de E. coli existe t'il ?"
Pour répondre à cette question, je peux me contenter du schème explicatif molécularo--mécaniste1. Le réseau existe parce que les protéines, ADN, métabolites interagissent ensemble selon les lois de la physique et de la chimie. Ces interactions créent un réseau non linéaire à partir duquel, on le croit2, émerge la vie.
Mais je peux aussi répondre à cette question en utilisant le schème explicatif darwinien. Le réseau existe parce que l'algorithme évolutif a façonné le génome pendant plusieurs milliard d'années. L'environnement sélectionne les mutations bénéfiques c'est-à-dire celles qui confèrent aux individus une plus forte capacité à se reproduire. Or pour se reproduire, il faut déjà survivre et pour survivre, il faut pouvoir s'adapter à des changements dans l'environnement. La présence d'un réseau de régulation permet cette adaptation et confère donc un avantage sélectif aux individus qui le possèdent.
Ce chapitre sera découpé en 3 parties:
- la première partie rappelle quelques généralités concernant l'évolution.
- la seconde partie se focalise plus particulièrement sur le rôle de l'évolution dans l'apparition des réseaux de régulation.
- la troisième partie étudie les liens entre évolution et biologie synthétique.
Quelques généralités sur l'évolution
Contrairement à une idée répandue, la théorie de l'évolution n'est pas simple. La difficulté ne vient pas du pattern général. Elle vient du fait qu'en matière d'évolution, "le diable est dans les détails" et j'essaierai de le demontrer avec plusieurs exemples. Notez également que j'aborde moi-même ce sujet en tant que novice (mon école de pensée d'origine étant plutôt "molécularo--mécaniste" ). Mon exposé pourrait refléter une compréhension partielle voire erronée de la théorie de l'évolution. Il doit donc être lu avec prudence et sens critique.
Dans l'évolution, j'identifie différents thèmes qu'il convient de traiter correctement:
- les mutations
- le transfert horizontal de gènes
- la sélection naturelle
- la dérive génétique
- les contraintes évolutives
Les mutations
Nous l'avons vu, lors de la réplication de l'ADN, il arrive que l'ADN polymérase se trompe et insère le mauvais nucléotide. Il existe des mécanismes de correction d'erreurs mais il y a toujours des mutations qui y échappent. Au final, le taux de mutation moyen chez E. coli est de l'ordre de 10-9 mutations par paire de bases par génération. Par conséquent, sur une population de 1 milliard de bactéries, il y a statistiquement une bactérie mutante pour chacun des nucléotides. Comme le chromosome d' E. coli contient très approximativement 1 million de nucléotides. Sur une population de 1 milliard de bactéries, il y a un million de bactéries mutantes. Ainsi il y a en moyenne une bactérie sur mille qui possède une mutation. On voit donc que, dés lors que l'on dépasse mille bactéries dans une flasque, la population n'est plus génotypiquement homogène. Il y a une distribution des différents génotypes. La plupart des mutations sont soit neutres (sans conséquence directe) soit délétères (elle diminue le fitness de la bactérie). Quelques unes apportent un avantage, c'est celles-là qui seront sélectionnées. Selon le paradigme actuel, les mutations se produisent de manière strictement aléatoire lors des erreurs de réplication. Par conséquent, la bactérie ne peut pas choisir le nucléotide à muter en fonction de l'environnement (Lamarckisme). En revanche, la bactérie peut jouer sur le taux de mutation via le contrôle des mécanismes de réparation (Barrick et al. 2009). Dans un environnement hostile par exemple, avoir un taux de mutation plus élevé peut permettre de "fouiller" plus vite les possibilités d'adaptation mais le prix à payer c'est un grand nombre de mutants, eux, beaucoup moins adaptés.
Le transfert horizontal de gènes
Le transfert horizontal de gènes est un processus dans lequel un organisme intègre du matériel génétique provenant d'un autre organisme sans en être le descendant. Chez les bactéries, il survient de 3 manières: par conjugaison, par transduction ou par transformation. La conjugaison est le transfert de matériel génétique (souvent un plasmide) par contact direct de cellule à cellule. A l'inverse, la transduction et la transformation s'effectuent sans contact intercellulaire. La transduction permet la récupération d'ADN provenant d'un autre organisme par l'intermédiaire d'un virus (chez les bactéries, on parle de bactériophage) tandis que la transformation est la récupération d'ADN extracellulaire présent dans l'environnement. On estime qu'au moins 18% du génome de E. coli provient de transfert horizontal de gènes (Lawrence and Ochman 1998). Le transfert horizontal a sans doute joué un rôle important dans l'évolution des réseaux de régulation (McAdams et al. 2004; Price et al. 2008) en fournissant aux bactéries des modules complets dont les paramètres peuvent être facilement "tunés" avec des mutations. Notez que les algorithmes génétiques informatiques sont de redoutables heuristiques qui permettent de résoudre des problèmes d'optimisation difficiles. Ils tirent en partie leur puissance de la capacité à combiner ensemble de "bons" paramètres par transfert horizontal de gènes (équivalent du crossing-over).
La sélection naturelle
Dés lors que la population n'est plus génotypiquement homogène, les différents génotypes se retrouvent en compétition pour les ressources de l'environnement. La sélection naturelle explique comment l'environnement influe sur l'évolution des génomes en sélectionnant les individus les plus adaptés. De manière plus formelle, "La sélection naturelle désigne le fait que les traits qui favorisent la survie et la reproduction, voient leur fréquence s'accroître d'une génération à l'autre. Cela découle logiquement du fait que les porteurs de ces traits ont plus de descendants, et aussi que ces derniers portent ces traits" 3 . Derrière l'apparente simplicité du concept de sélection naturelle se cache le débat le plus intense et le plus passionné de toute la philosophie de la biologie au cours des quarante dernières années: le débat sur les unités de sélection ---débat toujours ouvert aujourd'hui en 2011---.
C'est l'ouvrage controversé de Richard Dawkins Le gène égoïste publié en 1976, vendu à plus d'un million d'exemplaires et traduit dans 25 langues qui "enflamme" le débat parmi les biologistes et philosophes de la biologie (dont les principaux représentants sont Dawkins, Gould, Lewontin, Maynard-Smith, Mayr et Hull). La proposition de Dawkins tient en une phrase: bien que l'on ait cru jusqu'ici que la bonne unité de sélection était l'individu, en réalité la bonne unité de sélection est le gène. Selon lui, les gènes qui se sont imposés sont ceux qui provoquent des effets qui servent leurs intérêts propres (c'est-à-dire de continuer à se reproduire) et pas forcément les intérêts de l'individu. Citons l'exemple du comportement instinctif (et donc dicté par leurs gènes) de ces araignées mâles qui tentent de se reproduire tout en s'exposant au cannibalisme de la femelle. Cette vision des choses explique aussi l'altruisme au niveau des individus. Par exemple, quand un individu se sacrifie pour protéger la vie d'un membre de sa famille, il agit dans l'intérêt de ses propres gènes.
Dans le deuxième chapitre sur l'évolution, je formulerai une critique de la notion de gène égoïste et reviendrai plus en détail sur le débat des unités de sélection. De manière très simpliste, notez que les opposants à l'idée que le gène est la seule unité de sélection défendent une vision plus hiérarchique. Selon eux, la sélection naturelle peut s'opérer à différents niveaux (gènes, génomes, cellules, organismes, espèces, populations, écosystèmes...).
La dérive génétique
La dérive génétique est l'évolution d'une population ou d'une espèce causée par des phénomènes aléatoires, impossibles à prévoir. La dérive peut avoir pour conséquence la fixation de mutation délétère ou au contraire la perte de mutation bénéfique. Elle est causée par le biais d'échantillonnage lié au fait que les effectifs d'une population sont toujours finis. Par conséquent, son effet est plus fort sur les petites populations mais agit également sur les grandes.
Pour illustrer la différence essentielle entre dérive génétique et sélection naturelle, je vais décrire deux expériences d'évolution expérimentale très proches mais aux conséquences opposées.
Dans la première expérience, on part d'une colonie isolée que l'on met dans un milieu nutritif liquide bien défini. Les bactéries se divisent de manière exponentielle et une fois qu'elles atteignent la phase stationnaire (par exemple 107 bactéries), on les dilue 100 fois (105 bactéries) dans du nouveau milieu frais de même composition. Et on fait cela tous les jours ce qui aboutit à des milliers de génération4. Les bactéries qui ont des mutations bénéfiques se divisent plus vite. Par conséquent, elles transmettent plus souvent leurs mutations à leur descendance et petit à petit cette dernière devient majoritaire. Ce que l'on observera c'est une augmentation du fitness au cours des générations successives. Les bactéries s'adaptent de mieux en mieux à leur environnement. C'est l'effet de la sélection naturelle.
Maintenant faisons une autre expérience. On part d'une colonie isolée que l'on strie sur boite (milieu solide nutritif bien défini) ce qui permet d'isoler des bactéries à différents endroits sur la boite. Pendant la nuit, les bactéries isolées vont se diviser une fois, deux fois, 4 fois de manière exponentielle et le lendemain matin, on observera des colonies rondes (des millions de bactéries) provenant initialement de bactéries uniques. On re-strie une colonie sur boite pour isoler à nouveau des bactéries et ainsi de suite tous les jours pendant des milliers de générations. Contrairement à l'expérience précédente, ce que l'on observera c'est une diminution du fitness au cours des générations successives. Les colonies successives deviennent de moins en moins adaptées. Autrement dit, les bactéries se divisent de moins en moins vite. C'est l'effet de la dérive génétique. Pourtant, cette expérience ressemble beaucoup à l'expérience précédente: Pourquoi observons-nous alors le résultat inverse ?
La différence essentielle vient de l'échantillonnage très différent. Dans la première expérience, on dilue 100 fois les bactéries dans du milieu frais tous les jours. Les 105 bactéries que l'on récupère sont un panel représentatif des 107 bactéries ayant participé à la compétition et donc ayant subi les effets de la sélection naturelle. Au contraire, dans la deuxième expérience, le passage de la colonie à la bactérie unique crée une dilution bien plus forte, un énorme goulot d'étranglement. En sélectionnant une colonie provenant d'une bactérie unique, l'expérimentateur ne "capitalise" pas les effets de la sélection naturelle. Au fil des cycles, les colonies successives accumulent des mutations délétères (statistiquement beaucoup plus nombreuses que les mutations bénéfiques) ce qui explique la diminution du fitness au cours des générations.
Les contraintes évolutives
Selon Stephen J. Gould, l'évolution est contingente (possibilité qu'une chose arrive ou n'arrive pas) ce qui signifie que si l'on devait "rejouer" les 4 milliards d'années d'évolution depuis l'apparition de la vie, il est peu probable que l'homme réapparaitrait. L'apparition de l'homme n'est pas nécessaire. En évolution, "tout n'est pas mieux dans le meilleur des mondes" ce qui signifie que Pangloss5 et les "adaptationnistes" se trompent. L'évolution contient une part de hasard et la sélection naturelle ne saurait aboutir à la perfection, à une adaptation optimale. L'incapacité qu'à la sélection naturelle à conduire à l'optimisation idéale est due à la présence de contraintes que Maynard-Smith définit comme cela:
"Les contraintes développementales peuvent se définir comme un biais dans la production de phénotypes variants, ou une limitation de la variabilité phénotypique, causée par la structure, le caractère, la composition ou la dynamique du système développemental." (Maynard-Smith 1985) 6
Penser en termes de contraintes aide à s'affranchir d'une des dérives du darwinisme qui consiste à ne raisonner qu'en termes de fonctions c'est à dire d'utilité immédiate des adaptations. En effet, d'autres explications sont possibles pour expliquer l'existence des structures biologiques. Pour comprendre cela, je vais rappeler l'exemple très classique (et polémique) des spandrels (en réalité des pendentifs) que l'on doit à Gould et Lewontin (Gould and Lewontin 1979). Ces derniers notent que les espaces entre les arches qui supportent le toit de la basilique St Marc à Venise sont magnifiquement décorés. Les auteurs soutiennent que bien que ces espaces triangulaires aient été admirablement bien utilisés, ils ne sont pourtant que des sous produits architecturaux vis-à-vis de l'emploi des arches soutenant le dôme du toit. En d'autres termes, leur présence n'est pas due initialement à leur utilisation artistique. La leçon à retenir est simple: lorsque l'on se trouve face à un trait particulier, on ne devrait pas conclure que sa fonction particulière (ici artistique) est la raison pour laquelle le trait est présent. On retiendra donc que chacune des caractéristiques d'un organisme n'est pas toujours le produit direct de la sélection.

C'est dans la lignée de cette intervention sur les spandrels qu'un nouveau terme apparait en 1982: l'exaptation (Gould and Vrba 1982). Gould et Vrba reprennent l'idée que la sélection qui s'exerce sur une structure et la maintient en vue d'une "utilité" actuelle n'est pas toujours celle qui explique son apparition originelle. Le rôle passé et le rôle présent de la sélection pour les caractéristiques des organismes peuvent être tout à fait différents. Nietzsche, qui s'intéressait beaucoup à la biologie, avait déjà décrit ce concept il y a plus d'un siècle :
"Tout événement du monde organique est une manière de subjuguer, de dominer et toute subjugation, toute domination, à leur tour équivalent à une nouvelle interprétation, à un accommodement, où le sens et le but antérieur s'obscurciront nécessairement et même disparaitront tout à fait. On a beau comprendre l'utilité d'un organe physiologique (ou d'une institution juridique, d'une coutume sociale, d'un usage politique, ou encore d'une forme artistique ou d'un culte religieux), on n'a pour autant rien compris encore à sa naissance: pour gênant et désagréable que cela soit à de vieilles oreilles - de tout temps en effet on a cru que la finalité démontrable, l'utilisé d'une chose, d'une forme, d'une institution, était aussi la cause de leur naissance: l'oeil aurait été fait pour voir, la main pour saisir.[...] Tout but, toute utilité ne sont cependant que des symptômes indiquant qu'une volonté de puissance s'est emparée de quelque chose de moins puissant qu'elle et lui a de son propre chef imprimé le sens d'une fonction ; et toute l'histoire d'une chose, d'un organe, d'un usage peut être ainsi une chaîne continue d'interprétations et d'adaptations toujours nouvelles, dont les causes ne sont même pas nécessairement en rapport les unes avec les autres, mais peuvent se succéder et se remplacer les unes les autres de façon purement accidentelle." 7
Un très bel exemple d'exaptation a été découvert en 2009. Certains mammifères nocturnes utilisent une stratégie peu commune pour la vision nocturne (pour détecter des photons uniques). Quand la lumière s'estompe, la chromatine des noyaux de leurs cellules en bâtonnet change la transcription d'une part mais se réorganise aussi de telle manière à se comporter comme une lentille. Cela permet de focaliser les photons sur les récepteurs photosensibles là où le signal lumineux est ensuite traduit en influx nerveux. Ainsi, la quantité de lumière détectée est augmentée, un avantage certain dans des conditions de faible luminosité. On comprend bien que cette nouvelle fonction qu'a l'ADN (lentille) n'a rien avoir avec son rôle de support du programme génétique. Il serait stupide de dire que l'évolution a inventé l'ADN en cherchant à optimiser la vision nocturne. Ici l'arche qui soutient le dôme c'est l'ADN support de l'information génétique. A partir de là, il y a de nouveaux espaces qui sont contraints (les spandrels) et que l'évolution utilise pour faire apparaitre de nouvelles fonctionnalités (l'ADN comme lentille).
Evolution et réseau de régulation
Pourquoi l'évolution aurait t'elle sélectionné la présence d'un réseau plutôt que son absence ?
Nous savons que les gènes sont modifiés par l'évolution. Mais les promoteurs, RBS, facteurs de transcription eux aussi subissent les effets des mutations ce qui fait que le réseau est évidemment lui aussi finement "tuné" par l'évolution. Quel peut-être l'intérêt pour une bactérie de posséder un réseau de régulation couteux métaboliquement alors qu'elle pourrait se contenter de garder constante et optimale l'activité du promoteur de chaque gène ? Et si la bactérie a un intérêt dans la possession d'un réseau de régulation, ce dernier est-il toujours optimal ? Optimisé par l'évolution ?
Dés lors que l'on est confronté à un problème d'optimisation, il faut pouvoir définir précisément la fonction à optimiser ce qui n'est pas toujours évident car nous ne la connaissons pas toujours: par exemple quelle est la fonction à optimiser des cellules d'un tissu particulier, par exemple la peau? On ne sait pas. Avec les bactéries, les choses sont plus simples: dans le cas théorique où les bactéries poussent dans un environnement constant constamment réapprovisionné, on peut utiliser le taux de croissance comme fonction à optimiser (le fitness). En effet, dans ce cas, la pression évolutive maximise le taux de croissance. Pour calculer cette fonction de fitness, il faut réfléchir en termes de coût et de bénéfice. Nous allons voir comment la sélection naturelle peut:
- adapter le niveau d'expression d'un gène.
- choisir de réguler ou de ne pas réguler l'expression d'un gène.
- choisir d'utiliser un "motif" complexe pour réguler l'expression d'un gène par rapport à une simple activation/répression.
Adapter le niveau d'expression d'un gène
Qu'est-ce qui détermine le niveau optimal d'expression du gène lacZ codant pour la β-galactosidase, l'enzyme nécessaire pour utiliser le lactose comme source d'énergie? Le fitness f de la bactérie dépendra du bénéfice b qu'elle tire de la présence de β-galactosidase auquel il faut soustraire le coût c de production de cette enzyme.
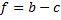
La courbe du taux de croissance en fonction de la concentration de β-galactosidase a une forme de cloche. Quand la concentration de β-galactosidase est trop faible (non optimale), la bactérie dégrade le lactose trop lentement et pousse donc également lentement. Si on augmente la concentration, on finit par atteindre le maximum de la courbe en cloche: la concentration de β-galactosidase est optimale et par conséquent, l'ajout de nouvelles enzymes n'améliore plus le taux de croissance. Au contraire, le coût métabolique (transcription et traduction) ainsi que la monopolisation de ressources (acides aminés) qui pourraient être utilisées pour d'autres protéines finit par diminuer le taux de croissance.
L'équipe d'Uri Alon8 a mené une expérience d'évolution expérimentale. Ils sont partis de plusieurs tubes, chacun avec une concentration différente de lactose, dans lesquels ils ont fait évoluer indépendamment une souche sauvage d'E. coli. Ils ont pu montrer qu'en l'espace de quelques centaines de générations, des mutations apparaissent permettant aux bactéries d'adapter leur concentration de β-galactosidase à un niveau optimal: forte concentration de β-galactosidase quand il y a une forte concentration de lactose et inversement. Bien sûr, la population la mieux adaptée prenait très rapidement le dessus sur les autres (Dekel and Alon 2005).
Réguler ou ne pas réguler
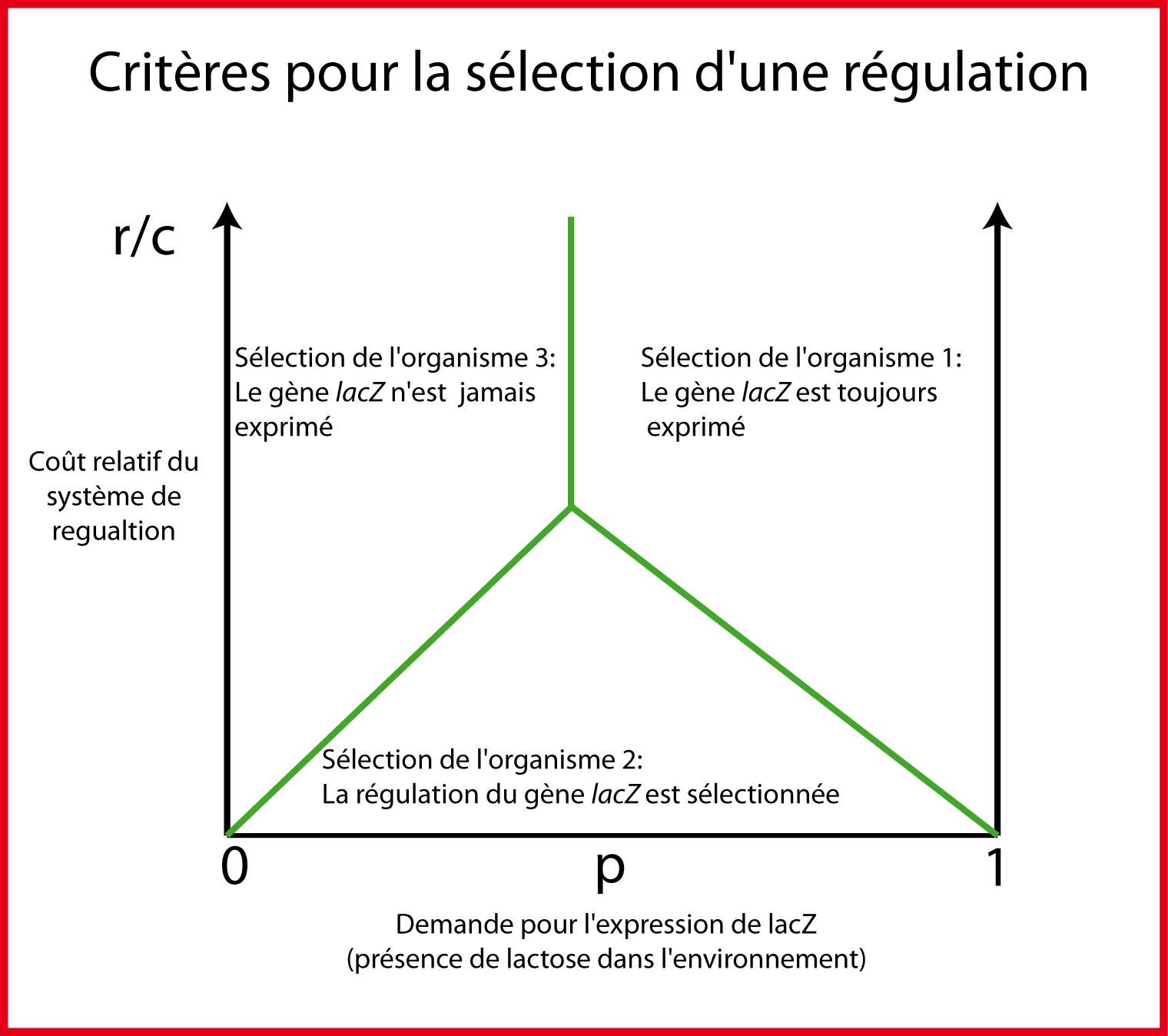
Dans cette partie, on va étudier pourquoi certains gènes sont régulés alors que d'autres sont exprimés de manière constitutive. Il nous faut tout d'abord considérer, non plus un environnement constant, mais un environnement variable. Par exemple, dans cet environnement variable, il y a parfois du lactose et parfois il n'y en a pas. Or le gène lacZ n'apporte un bénéfice direct au fitness que lorsque le lactose est présent. La présence d'une régulation signifie que le gène lacZ n'est produit que lorsque le lactose est présent. Mais cette régulation a un coût: le coût de sa production et de sa maintenance. Ainsi la régulation n'aura un intérêt que si son bénéfice est supérieur à son coût. Pour analyser la stratégie optimale, nous allons comparer trois organismes avec 3 designs différents pour la régulation du gène lacZ.
Appelons p la fraction du temps où le lactose est présent dans le milieu. p représente la demande pour l'expression de lacZ. Le reste du temps (1-p), l'expression de lacZ est inutile.
Dans l'organisme 1, lacZ est exprimé de manière constitutive (sans régulation). Le bénéfice b n'a lieu que lors de la fraction p du temps, quand le lactose est présent. Son fitness f1 est donc:
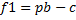
L'organisme 2 possède une régulation: il n'exprime lacZ qu'en présence de lactose. De cette manière, l'organisme économise la production non nécessaire lorsque le lactose est absent: il ne paye donc le coût c qu'une fraction du temps p. Cependant il faut payer le prix r pour produire et maintenir le système de régulation.
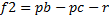
Enfin, le troisième organisme ne produit pas du tout lacZ. Le fitness f3 sur lactose est égal à zéro: c'est le niveau de référence où il n'y a ni coût ni bénéfice.
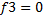
La régulation est sélectionnée par l'évolution lorsque l'organisme 2 a le plus haut fitness c'est-à-dire
lorsque 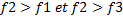 . Cela nous amène aux deux inégalités
suivantes:
. Cela nous amène aux deux inégalités
suivantes:
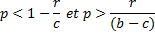
Ces inégalités relient une propriété de l'environnement (la demande pour l'expression de lacZ correspondant à la fraction du temps où le lactose est présent) aux paramètres de coût et de bénéfice de l'expression de lacZ ainsi que son système de régulation. Pour chaque valeur de p, ces équations nous disent si le système de régulation sera ou non sélectionné par rapport aux autres designs plus simples. La gamme des environnements dans lesquels chacun des 3 designs est optimal est montrée dans la figure 2.
Sélection d'un motif
Nous avons vu dans le chapitre sur les systèmes dynamiques qu'il existe certains motifs (des circuits élémentaires récurrents) qui apparaissent statistiquement plus souvent dans le réseau de régulation génique de E. coli par rapport à un réseau strictement aléatoire. Il semble donc que ces motifs aient été sélectionnés par l'évolution. Nous avons vu que l'un de ces motifs, le feed forward loop, agit comme un détecteur de persistance.
Dans un environnement où la présence et l'absence de lactose varie au cours du temps, il peut y avoir des petits pulses de lactose, par exemple de quelques minutes, et des pulses plus longs où le lactose reste présent pendant plusieurs heures. Si le système de régulation du lactose déclenche l'expression de lacZ lors des petits pulses, le coût risque d'être plus important que le bénéfice car peut être que lorsque les premières molécules de β-galactosidase arriveront, le lactose aura déjà disparu. La présence d'un motif régulateur plus complexe (le feed forward loop ou "FFL") filtre ces petits pulses et empêche qu'ils ne déclenchent l'induction de lacZ. Il en résulte un gain de fitness potentiel. En utilisant, la même stratégie d'étude des coûts--bénéfices, on montre facilement qu'il existe un espace dans le diagramme des phases où le Feed forward loop est sélectionné par rapport à une régulation simple (Dekel et al. 2005).
Selon l'équipe d'Uri Alon, la présence d'un motif donné (par exemple le FFL) et donc sa sélection par l'évolution, signifie qu'il est le mode de régulation optimal par rapport à une condition environnementale donnée. Par exemple, si la régulation de lacZ est contrôlée par un FFL, cela signifie qu'il doit exister de nombreux pulses de lactose dans l'environnement qui nécessitent d'être filtrés. Autrement dit, on peut imaginer faire de l'écologie inversée et déduire des informations de l'environnement que rencontre une bactérie en fonction de son réseau de régulation génique. Cela est basé sur l'idée qu'un circuit optimal contient/est un modèle interne de l'environnement.
La loi de la demande
Pour continuer sur cette idée, vous avez peut-être remarqué que l'activateur et le répresseur transcriptionnel ont exactement le même but: n'exprimer un gène qu'en présence d'un signal donné. En effet, le signal peut activer un activateur (arabinose--AraC) ou inactiver un répresseur (lactose9 --LacI). Parmi ces deux systèmes équivalents, l'évolution en choisit un. Mais y-a-t-il des règles qui gouvernent cette sélection ou est-ce que l'évolution choisit au hasard répression ou activation ?
Cette question a été soulevée par Savageau qui a montré que le mode de contrôle est corrélé à la demande (fraction du temps où l'expression du gène est nécessaire). Les gènes qui sont souvent "demandés " tendent à être contrôlés par une régulation positive. A l'inverse, les gènes dont le produit est peu demandé, tendent à être contrôlés négativement. Vous voyez donc qu'avec cette règle, en connaissant le mode de régulation d'une enzyme (positive ou négative), vous pouvez inférer l'abondance ou la rareté du substrat correspondant de l'environnement "sauvage" de la bactérie.
L'équipe d'Uri Alon a proposé une explication à ce phénomène. Elle repose sur le fait que quand un site de fixation d'un régulateur sur l'ADN est "libre", il est plus exposé à des fixations non spécifiques alors que le site sur lequel est fixé le bon régulateur est, lui, mieux protégé des erreurs. En effet, ces erreurs causées par une fixation d'un régulateur non spécifique peuvent changer l'expression du gène (de manière non voulue) et diminuer le fitness de la bactérie à cause du coût inutile que cela entraine.
Considérons maintenant que l'expression de lacZ est contrôlée par un activateur. Si le lactose est présent très souvent, alors l'activateur est très souvent fixé sur le promoteur ce qui protège des erreurs de fixation non spécifique réduisant le fitness. A l'inverse, si le lactose est très rare, l'activateur ne sera que rarement fixé au promoteur. Ce dernier sera alors bien plus exposé aux erreurs de fixation non spécifique ce qui réduira le fitness de la bactérie. Dans ce deuxième cas, la meilleure stratégie consiste à réguler l'expression de lacZ avec un répresseur de telle manière que le site ADN soit protégé la majorité du temps.
Imaginez maintenant que pendant des milliers d'années (ou de générations quand on parle en temps évolutif), une bactérie ait été exposée très souvent au lactose. Par conséquent, elle régule positivement l'expression de lacZ. Imaginez maintenant que, suite à un changement climatique par exemple, le lactose devienne plutôt rare. L'activateur étant moins souvent fixé sur le promoteur, il va y avoir une augmentation des fixations non spécifiques qui déclenchent l'expression de lacZ inutilement. Est-ce que le coût sur le fitness généré peut avoir pour conséquence d'inverser, au fil du temps évolutif, le mode de régulation (de activateur à répresseur) ? Et bien il semble que la réponse soit oui (Shinar et al. 2006): des mutants avec le mode de régulation inverse finissent par être sélectionnés.
Apprentissage associatif
Nous avons vu dans les exemples ci-dessus qu'il peut exister une corrélation entre la topologie et les paramètres d'un réseau de régulation d'une part et certaines conditions environnementales d'autre part. Avant de donner un nouvel exemple de la puissance de l'évolution pour modeler un réseau de régulation reflétant l'environnement extérieur, je souhaite vous ramener au lycée en vous rappelant la petite expérience de Pavlov. Le chien de ce dernier avait "associé" le son d'une sonnette avec l'arrivée de la viande. Il suffisait ensuite d'agiter la sonnette (sans forcement apporter la viande) pour que le chien se mette à saliver. Evidement, ce type d'apprentissage associatif passe par le système nerveux. Et bien, on pense que les bactéries sont capables d'apprentissage associatif via, non plus le système nerveux (qu'elles ne possèdent pas), mais via leur réseau de régulation.
Quand E. coli arrive dans notre bouche, elle va tout suite faire l'expérience d'une augmentation de température (37°C). Puis elle se retrouve dans le tractus gastro-intestinal où nous avons vu qu'elles sont en compétition avec de centaines d'autres espèces bactériennes (E. coli y est minoritaire: inférieur à 1%). Dans le tractus gastro-intestinal, la concentration en oxygène chute et les bactéries doivent "switcher" sur un programme de respiration anaérobie. Les bactéries peuvent détecter cette chute et adapter leur expression génique pour y faire face. Mais la compétition est rude dans les intestins et une bactérie qui "switch" en mode anaérobie dés l'augmentation de température dans la bouche, sera tout de suite "prête" à affronter les conditions anaérobiques des intestins. Il n'y aura pas le "délai" classique de "détection--expression". Le gain de fitness est probablement suffisant pour permettre la sélection de cette bactérie. Ainsi, l'évolution détecte la corrélation [augmentation de la température-- diminution de la pression partielle en oxygène] et l'imprime dans le réseau de régulation de la bactérie (Tagkopoulos et al. 2008). Ces résultats laissent donc bien supposer que l'on peut travailler de manière inverse dans un laboratoire (reverse-ingénierie): l'étude des corrélations (temporelles ou pas) présentes dans l'expression génique d'un organisme pourrait nous aider à reconstituer les corrélations d'ordre écologiques que vit ce même organisme.
Le reflet du miroir
La métaphore du "reflet du miroir" entre environnement et réseau de régulation/génome semble encore appropriée avec l'exemple ci-dessous. Une des questions essentielles à l'étude des réseaux de régulation est: sont-ils modulaires ? Et si oui, comment et pourquoi l'évolution a-t-elle sélectionné la modularité ? Kashtan a fait évoluer des réseaux de régulation in silico en utilisant des algorithmes évolutifs classiques. Il a pu démontrer que "ses" réseaux deviennent modulaires s'il change l'environnement de manière modulaire. Chaque environnement correspond en fait à un but évolutif (une fonction de fitness) et chacun de ces buts évolutifs est composé de sous buts (Kashtan and Alon 2005). Selon Kashtan, les variations dans l'environnement pourraient accélérer l'évolution: plus il y aurait de changements dans l'environnement, plus une bactérie "naviguerait" facilement dans le paysage adaptatif à la recherche de l'optimum global (Kashtan et al. 2007). Kashtan n'a pas manqué de voir que cette manière de procéder pourrait significativement améliorer les algorithmes évolutifs (informatiques) d'optimisation.
Evolution et biologie synthétique
Depuis plusieurs milliers d'années déjà, l'homme utilise l'évolution pour sélectionner des traits qui l'intéressent chez certaines espèces. L'exemple de sélection artificielle qui me semble le plus percutant c'est celui du loup et du chien. L'hypothèse communément admise chez les scientifiques est que le chien descend du loup. On pense que les chasseurs--cueilleurs adoptaient parfois des bébés loup. En grandissant, certains des louveteaux devenaient agressifs et étaient alors tués. Les loups au caractère "dominé" étaient ainsi sélectionnés pour leur absence d'agressivité, leur "bonne compagnie". L'homme en protégeant et nourrissant les loups (devenus chien) a créé un barrage de protection génétique contre les effets de la sélection naturelle (l'arche qui soutient le toit de la cathédrale). L'espace créé derrière ce barrage (les spandrels) est à l'origine de la très forte instabilité génétique du chien dont la conséquence est la multiplicité de races phénotypiquement très différentes. La fonction sélectionnée par l'homme, la "bonne compagnie" ne pourrait pas apparaitre seule dans la nature sans la présence de l'homme. Dit humoristiquement, si vous abandonnez un caniche royal en Sibérie, il est peu probable qu'il devienne le mâle "alpha" d'une meute de loup.

Avec cet exemple du chien, on voit que l'homme a construit (au fil des décennies/siècles) un organisme capable d'exécuter une fonction. La méthode est une méthode "à l'aveugle" qui utilise le principe de sélection artificielle: on ne garde que les individus qui s'approchent le plus de la fonction désirée et on les fait se reproduire entre eux. On est tellement habitué à cette manière de procéder que cela ne nous choque pas. Cela semble presque naturel. En biologie synthétique, les choses sont un peu différentes. On vient spécifiquement changer ce nucléotide là sur l'ADN pour transformer le molosse en caniche (j'exagère un peu). L'évolution change le même nucléotide mais sans fournir l'information. En ce début de 21ème siècle, pour le même résultat (obtenir telle fonction), la présence du hasard couplé à l'absence d'information rassure (dieu, la nature) alors que la connaissance de l'information (savoir quel nucléotide changer) apparaît diabolique.
Le but du biologiste synthétique consiste à construire un organisme, à contrôler une fonction en se passant de l'évolution. Le rêve serait de pouvoir, étant donnée une séquence nucléotidique, calculer la fonction (l'organisme) ou au contraire, étant donnée une fonction (l'organisme) calculer la (ou les) séquence nucléotidiques.
C'est exactement ce qu'a fait Michael Elowitz avec son plasmide repressilator (Elowitz and Leibler 2000): il combine des petites séquences nucléotidiques simples: 3 promoteurs et 3 gènes dont il connait la fonction pour créer une séquence nucléotidique plus compliquée qui code pour une fonction, elle aussi, plus complexe: des oscillations dans la concentration de protéines au cours du temps. Et ça a marché. Mais cette approche "plug and play" (empruntée à l'électronique) qui consiste à combiner ensemble des séquences nucléotidiques avec des fonctions simples pour générer des fonctions plus complexes montre très vite ses limites en biologie. La raison est simple: le niveau de standardisation de ces briques de base est très faible par rapport aux briques de base des microprocesseurs (les transistors). Le niveau d'incertitude et de méconnaissance est très important et pose donc très vite problème. Imaginez que vous combiniez quelques "biobriks" ensemble pour générer une fonction complexe et qu'une fois dans la cellule, la séquence ne génère pas le phénotype attendu ? Ce n'est peut être qu'un paramètre (par exemple la force d'un promoteur) qui mérite d'être ajusté ? Mais comment savoir quel nucléotide modifier? On peut faire du "rational design" et tenter de modifier un nucléotide donné par mutagénèse dirigée en espérant être en mesure de prédire correctement le résultat mais c'est une approche chronophage et donc risquée. Une solution consiste à réutiliser l'évolution (une dernière fois) pour tuner finement les paramètres, c'est-à-dire pour accroitre la connaissance/l'information sur la séquence en vue d'obtenir la meilleure correspondance fonction--séquence. On parle d'évolution dirigée.
Utiliser l'évolution pour "tuner" un réseau de régulation synthétique
Il existe plusieurs étapes majeures dans une expérience d'évolution dirigée. La diversité génétique est crée par mutagenèse aléatoire et/ou recombinaison in vitro de une ou plusieurs séquences parentes (DNA shuffling). Les séquences altérées sont ensuite clonées dans un plasmide pour expression du phénotype dans la souche hôte (par exemple E. coli). Les clones qui expriment la fonction améliorée sont identifiés par screening à haut débit. Les séquences qui encodent la fonction améliorée sont isolées pour le prochain cycle d'évolution dirigé.
Pour être clair, prenons l'exemple de l'amélioration de la GFP par évolution dirigée. Les chercheurs voulaient créer une GFP encore plus fluorescente. Ils ont crée une banque de séquences de GFP par de techniques de recombinaison in vitro. Les séquences sont clonées dans un plasmide pour expression chez E. coli. Il suffit ensuite d'identifier parmi les milliers de colonies, celles qui sont les plus fluorescentes puis de refaire ce cycle plusieurs fois en repartant des colonies sélectionnées. Par cette méthode, en 3 cycles seulement, les chercheurs ont réussi à obtenir une GFP 45 fois plus fluorescente (Crameri et al. 1996).
En appliquant cette méthode non plus à la GFP mais à un facteur de transcription, vous voyez que l'on peut modifier certains paramètres, par exemple diminuer ou augmenter son affinité à l'ADN.
Notez qu'il existe une alternative au screening qui consiste à utiliser la sélection pour identifier le phénotype recherché. Dans ce cas, on couple la fonction recherché à la survie cellulaire, par exemple en utilisant une cassette de résistance à un antibiotique. Cela permet d'assurer la pression évolutive nécessaire à la sélection.
Mais vous allez me dire "c'est génial, avec l'évolution dirigée, on peut tout faire !"
Par exemple, pourquoi ne pas partir d'un plasmide composé du même nucléotide, par exemple 10000 adénosines les unes à la suite des autres. On pourrait diriger l'évolution de cette séquence pour qu'elle encode une fonction donnée sélectionnée par l'expérimentateur, par exemple les oscillations dans la concentration de protéine.
Cela n'est évidemment pas possible pour une raison simple: l'espace des séquences à fouiller est astronomique. il y a 4 bases possibles (A,T,C,G) et 10000 pb soit 410000 séquences possibles. Le nombre de colonies que l'on peut raisonnablement "screener" ne peut guère dépasser le million. Mais même si on pouvait "screener" des milliards de milliards de séquences, on ne fouillerait qu'une infime partie de l'espace des séquences possibles. La probabilité de tomber au hasard sur la séquence du repressilator (séquence générant les oscillations) est quasi nulle.
La bonne démarche en faisant de l'évolution dirigée en biologie synthétique consiste à faire en sorte que la séquence qui encode la fonction recherchée ait une bonne probabilité d'être présente parmi les quelques milliers/millions de séquence "screenées". Pour cela, il y a deux solutions:
- si la séquence est longue (un gène de 1000 pb), il faut que la séquence recherchée ne soit qu'à quelques mutations (ou événements de recombinaison) de la séquence d'origine.
- si la séquence est courte, par exemple un RBS (ribosome binding site) de 7 pb et que l'on souhaite modifier l'efficacité de la traduction du gène en aval, on peut facilement tester l'ensemble des combinaisons 47 soit 16384 séquences car cela correspond à un nombre de colonies "screenable ". Cette approche a été utilisée pour "tuner" un petit réseau synthétique permettant à E. coli de décider d'envahir ou pas des cellules cancéreuses en fonction des conditions extérieurs (Anderson et al. 2006).
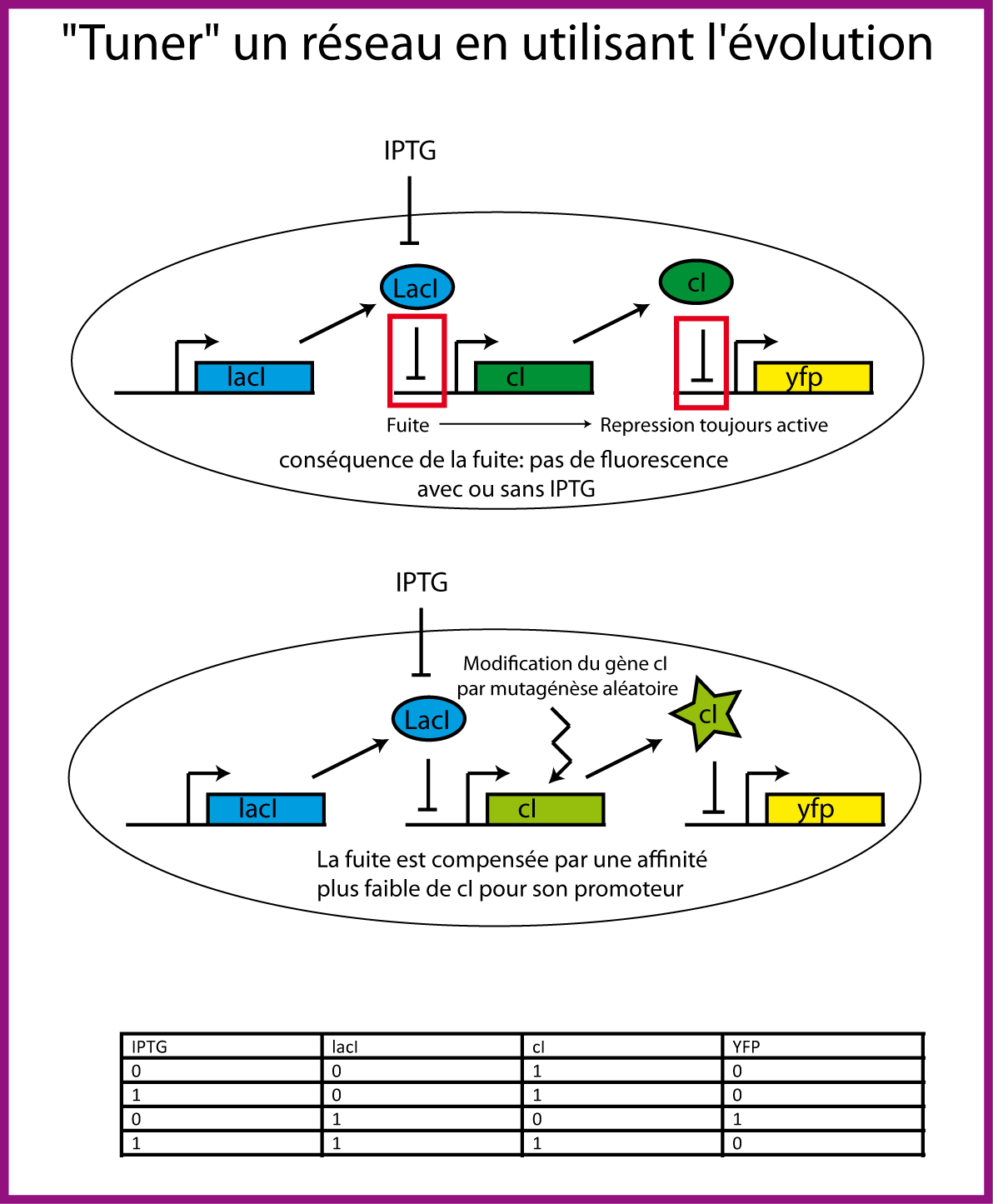
Nous avons vu que la démarche "plug and play" consistant à créer un petit réseau à partir de briques (séquences) de base est une approche qui montre très vite ses limites car la standardisation est moins aisée qu'en électronique. La bonne démarche est plutôt "plug, mutate and play". Le "plug" permet de réduire l'espace des séquences en fournissant la structure générale du système, le "mutate" se charge de modifier les quelques bases nécessaires pour obtenir les paramètres compatibles avec la fonction recherchée. Pour continuer l'analogie avec l'informatique, on peut dire que l'étape "mutate" correspond à l'étape de debugage d'un programme informatique: on modifie quelques caractères (parenthèses oubliées...) ou quelques segments pour que le code fonctionne. Un fois debuggé, il ne reste alors plus qu'à exécuter le programme (le "play" ). La correspondance séquence--fonction est alors optimale. Je vais maintenant décrire un bel exemple de la méthode "plug, mutate and play" issu des travaux de l'équipe de Frances H. Arnold (Yokobayashi et al. 2002).
Les chercheurs créent un petit réseau linéaire très simple où la protéine répresseur LacI réprime l'expression de la protéine répresseur cI qui, elle-même réprime l'expression de la yellow fluorescent protein (YFP). En présence de l'inducteur IPTG qui "inactive" allosteriquement la protéine LacI, cI devrait être exprimé et par conséquent YFP ne devrait pas l'être. A l'inverse, en absence d'ITPG, LacI empêche l'expression de cI et donc YFP devrait être exprimé. Le problème qui s'est posé, après avoir construit ce petit circuit simple, c'est que, avec ou sans IPTG, les colonies n'étaient jamais fluorescentes. Le problème venait d'une petite fuite dans le promoteur lac qui permettait, même en absence d'IPTG, une expression suffisante de cI pour réprimer l'expression de YFP. Les auteurs ont alors faire évoluer la protéine cI par mutagénèse aléatoire et ont "screené" les colonies montrant le phénotype attendu: fluorescence en absence d'IPTG et absence de fluorescence en présence d'IPTG. Ils ont réussi à isoler des mutants de la protéine cI dont l'affinité pour l'ADN est plus faible ce qui compense la fuite du promoteur lac et permet de retrouver le phénotype attendu. Les chercheurs démontrent ainsi que l'évolution dirigée peut aider à résoudre, in vivo, des problèmes complexes d'optimisation impliquant de nombreux paramètres. C'est donc une méthode puissante pour "tuner" finement un petit réseau (le debugger) et obtenir la propriété/la fonction désirée (Haseltine and Arnold 2007).
L'évolution peut-elle être un frein aux avancées en biologie synthétique ?
Mais une fois que l'on réussit à obtenir un réseau fonctionnel, une correspondance séquence--fonction optimale, le problème inverse se pose forcement. L'évolution ne s'arrête pas une fois que la bactérie est dans le fermenteur pour exécuter la fonction pour laquelle elle a été programmée. L'objectif de l'expérimentateur peut très vite s'opposer à l'objectif de l'évolution à savoir maximiser le fitness. La plupart des circuits synthétiques ont un coût métabolique or même quand ce coût est faible, il entraine une diminution du fitness par rapport à une souche sans circuit fonctionnel. Inexorablement, en seulement quelques jours (You et al. 2004), des mutants ayant inactivé le circuit apparaissent et finissent par devenir majoritaires (Arkin and Fletcher 2006).
Pour limiter les effets de l'évolution , on peut tenter de diminuer la fréquence d'apparition des mutations en surexprimant les protéines impliquées dans la réparation des erreurs (gène mutS, mutL) (Zhao and Winkler 2000) mais inexorablement des mutants mieux adaptés apparaitront. Car même si l'ADN polymérase est probablement la machine de copie la plus efficace sur terre, elle ne recopiera jamais l'ADN avec une fiabilité de 100%.
Réussir à isoler un circuit synthétique des effets de l'évolution ne sert pas seulement à protéger la fonction recherchée. Il faut aussi empêcher que le circuit n'évolue dans une direction que l'on ne contrôlerait pas et/ou qu'il ne se dissémine dans l'environnement contre notre volonté. Si on crée un "calculateur" synthétique, on ne veut pas que ce calculateur se balade d'organismes en organismes via les échanges de matériels génétiques, ne serait-ce que d'un point de vue éthique. Il y a un risque d'échappement que les biologistes combattent depuis longtemps en créant des stratégies de "confinement biologique". Il faut réussir à isoler le matériel génétique synthétique de l'environnement.
Une des sources majeures de changement évolutif dans la nature vient du transfert horizontal de gènes. Les 3 principales voies de transfert horizontal sont la conjugaison, la transduction et la transformation (décrites plus haut dans ce chapitre). Une méthode employée depuis longtemps consiste à supprimer dans la souche qui reçoit le "circuit synthétique" les gènes nécessaires à ces différents modes de transfert horizontal. Par exemple, on supprime les gènes de compétence (com), les gènes nécessaires à la recombinaison sur le chromosome (rec) ou bien les gènes indispensables à l'infection virale (Skerker et al. 2009).
La bioremediation est l'utilisation d'organisme vivants dont la fonction est de dépolluer un écosystème après une contamination toxique. Le microorganisme doit rendre l'écosystème tel qu'il était avant contamination (par exemple, en dégradant un composé toxique). Mais comment s'assurer que l'on contrôle l'action et l'invasion du microorganisme, que celui-ci ne modifie l'environnement que dans le sens voulu, et qu'une fois sa mission accomplie, il arrête d'agir ? Le problème c'est que l'évolution est parfaitement adaptée pour trouver la faille, pour casser toutes stratégies de "confinement" que l'on pourrait implémenter pour garder la souche sous contrôle. Il faut donc superposer plusieurs stratégies indépendantes de confinement de manière à ce que la probabilité d'échappement soit quasi nulle. Voici deux stratégies qui semblent assez robustes à l'évolution tout en étant faisables. Elles ne seront cependant pas simples à implémenter:
- le confinement nutritionnel consiste à rendre la souche dépendante d'une substance rare ou inconnue dans la nature pour survivre ou n'intervenant pas ou peu dans le vivant (Fluor, silice...) (Schmidt)
- le confinement sémantique consiste à utiliser un code génétique alternatif (orthogonal) de manière à ce que si par hasard l'ADN est transféré dans d'autres bactéries, la traduction ne produise que des protéines non fonctionnelles (Herdewijn and Marliere 2009).
On voit bien que dés lors que l'on implémente des fonctions avec l'ADN, il faut garder à l'esprit que ces fonctions sont susceptibles d'évoluer dans des directions non souhaitées et de se propager dans la nature. Contrôler l'évolution sera donc un passage obligé en biologie synthétique. Combattre un processus qui excelle à trouver des fuites, des failles dans la sécurité n'est pas chose aisé mais des solutions existent.
L'évolution de l'évolution
L'environnement ne peut pas changer directement (i.e sans passer par la sélection) la séquence du génome. Par exemple, si subitement la concentration du glucose dans le milieu augmente énormément, la bactérie ne peut pas détecter ce changement puis modifier spécifiquement tel nucléotide du promoteur du gène codant pour le transporteur du glucose dans le but d'augmenter fortement l'expression de ce dernier. On considère qu'il n'y a pas de lamarckisme: les mutations apparaissent toujours aléatoirement. On pense qu'il n'y a aucun biais statistique.
Mais le biologiste synthétique peut-il rendre ses lettres de noblesse à Jean-Baptiste de Lamarck ? Peut-on imaginer un système synthétique qui modifie spécifiquement l'ADN en fonction d'un stimulus externe ?
Les facteurs de transcription reconnaissent des sites spécifiques sur l'ADN et les systèmes de correction d'erreur sont capables de détecter un mésappariement sur l'ADN et de le corriger (Li 2008). En associant synthétiquement ces deux mécanismes, on peut imaginer créer une bactérie qui modifie son génome en fonction d'un stimulus externe.
Imaginez, par exemple, que l'on modifie le facteur de transcription AraC de telle manière à ce qu'en présence d'arabinose, il se fixe toujours en amont de l'operon araBAD mais n'active plus la transcription. A la place, il recrute la machinerie d'excision/réparation qui change un nucléotide (ce nucléotide là) sur le promoteur de telle manière à augmenter de manière "constitutive " l'expression de l'operon araBAD. Ce changement sera transmissible à la descendance. Evidemment, cela passe par la connaissance a priori dont dispose l'homme (il faut savoir quel nucléotide modifier). Mais l'exemple ci-dessus ne représente que le cas simple. La connaissance a priori des nucléotides à modifier spécifiquement en fonction des conditions extérieurs pourrait nous permettre de créer des réseaux extrêmement complexes chargés de "modeler" le génome en temps réel en fonction des conditions environnementales. De quoi faire se retourner Darwin dans sa tombe n'est-ce pas ?
Mais s'il semble relativement simple de faire du lamarckisme, pourquoi la nature n'a-t-elle pas choisi cette voie ?
Je vois deux arguments:
- modifier quelques nucléotides en fonction du milieu extérieur semble faisable mais contrôler l'ensemble des nucléotides du génome semble beaucoup plus complexe car les mécanismes chargés de contrôler la modification du génome en fonction de l'environnement sont eux aussi codés dans le génome et donc soumis à modification. Très vite, on perd l'intuition des conséquences potentielles. Il est possible que ces régulations lamarkiennes soient tout simplement théoriquement inimplementables à grande échelle.
- La nature ne dispose pas de l'information a priori (celle justement dont on dispose): elle ne sait pas que la modification de tel nucléotide induira telle conséquence sur le phénotype. En fait, l'homme a une idée partielle du paysage adaptatif de la bactérie qu'il utilise pour permettre à la bactérie de faire du lamarkisme.
Selon moi, plus on aura une connaissance précise a priori du paysage adaptatif, plus on pourra programmer des fonctions lamarckiennes. Mais j'avoue ne pas savoir si cela a un quelconque intérêt par rapport à un simple réseau de régulation. Il faudrait y réfléchir plus longuement.
J'ai appelé, un peu pompeusement d'ailleurs, le titre de ce paragraphe "évolution de l'évolution". L'idée était que les fonctions lamarckiennes que je propose d'implémenter synthétiquement représentent une évolution de l'évolution qui passe par l'invention du cerveau humain et sa compréhension des mécanismes moléculaires à l'origine de l'évolution. Mais j'avoue ne plus trop savoir si le titre est adéquat car je suis gêné par le fait que cette évolution de l'évolution passe par une connaissance a priori or le principe de l'évolution consiste justement à fouiller un paysage inconnu.
Bibliographie
- Anderson, J. C., E. J. Clarke, et al. (2006). "Environmentally controlled invasion of cancer cells by engineered bacteria." J Mol Biol 355(4): 619-27.
- Arkin, A. P. and D. A. Fletcher (2006). "Fast, cheap and somewhat in control." Genome Biol 7(8): 114.
- Barrick, J. E., D. S. Yu, et al. (2009). "Genome evolution and adaptation in a long-term experiment with Escherichia coli." Nature 461(7268): 1243-7.
- Crameri, A., E. A. Whitehorn, et al. (1996). "Improved green fluorescent protein by molecular evolution using DNA shuffling." Nat Biotechnol 14(3): 315-9.
- Dekel, E. and U. Alon (2005). "Optimality and evolutionary tuning of the expression level of a protein." Nature 436(7050): 588-92.
- Dekel, E., S. Mangan, et al. (2005). "Environmental selection of the feed-forward loop circuit in gene-regulation networks."
- Elowitz, M. B. and S. Leibler (2000). "A synthetic oscillatory network of transcriptional regulators." Nature 403(6767): 335-8.
- Gould, S. J. and R. C. Lewontin (1979). "The spandrels of San Marco and the Panglossian paradigm: a critique of the adaptationist programme." Proc R Soc Lond B Biol Sci 205(1161): 581-98.
- Gould, S. J. and E. Vrba (1982). "Exaptation; a missing term in the science of form " Paleobiology.
- Haseltine, E. L. and F. H. Arnold (2007). "Synthetic gene circuits: design with directed evolution." Annu Rev Biophys Biomol Struct 36: 1-19.
- Herdewijn, P. and P. Marliere (2009). "Toward safe genetically modified organisms through the chemical diversification of nucleic acids." Chem Biodivers 6(6): 791-808.
- Kashtan, N. and U. Alon (2005). "Spontaneous evolution of modularity and network motifs." Proc Natl Acad Sci U S A 102(39): 13773-8.
- Kashtan, N., E. Noor, et al. (2007). "Varying environments can speed up evolution." Proc Natl Acad Sci U S A 104(34): 13711-6.
- Lawrence, J. G. and H. Ochman (1998). "Molecular archaeology of the Escherichia coli genome." Proc Natl Acad Sci U S A 95(16): 9413-7.
- Li, G. M. (2008). "Mechanisms and functions of DNA mismatch repair." Cell Res 18(1): 85-98.
- Maynard-Smith, J., R. Burian, S. Kauffman, P. Alberch, J. Campbell, B. Goodwin, R. Lande, D. Raup, and L. Wolpert. (1985). "Developmental constraints and evolution. ." The Quarterly Review of Biology.
- McAdams, H. H., B. Srinivasan, et al. (2004). "The evolution of genetic regulatory systems in bacteria." Nat Rev Genet 5(3): 169-78.
- Price, M. N., P. S. Dehal, et al. (2008). "Horizontal gene transfer and the evolution of transcriptional regulation in Escherichia coli." Genome Biol 9(1): R4.
- Schmidt, M. "Xenobiology: a new form of life as the ultimate biosafety tool." Bioessays 32(4): 322-31.
- Shinar, G., E. Dekel, et al. (2006). "Rules for biological regulation based on error minimization." Proc Natl Acad Sci U S A 103(11): 3999-4004.
- Skerker, J. M., J. B. Lucks, et al. (2009). "Evolution, ecology and the engineered organism: lessons for synthetic biology." Genome Biol 10(11): 114.
- Tagkopoulos, I., Y. C. Liu, et al. (2008). "Predictive behavior within microbial genetic networks." Science 320(5881): 1313-7.
- Yokobayashi, Y., R. Weiss, et al. (2002). "Directed evolution of a genetic circuit." Proc Natl Acad Sci U S A 99(26): 16587-91.
- You, L., R. S. Cox, 3rd, et al. (2004). "Programmed population control by cell-cell communication and regulated killing." Nature 428(6985): 868-71.
- Zhao, J. and M. E. Winkler (2000). "Reduction of GC --> TA transversion mutation by overexpression of MutS in Escherichia coli K-12." J Bacteriol 182(17): 5025-8.
Notes de bas de page
- Voir la fin du chapitre 1, Michel Morange , Les secrets du vivant , La découverte.
- Lorsque l'on parle d'émergence, il faut rester prudent car cette notion ne respecte pas l'impératif de rigueur scientifique tel que défini par l'épistémologie institutionnelle positiviste.
- Wikipedia, article sur la "sélection naturelle".
- Cette expérience a été initié par Richard Lenski il y a quelques décennies et est toujours en cours aujourd'hui. Une des équipes (dirigé par le Pr. Dominique Schneider) de mon laboratoire travaille sur les données issues de ces expériences
- Pangloss est le précepteur de Candide dans le conte philosophique de Voltaire (1759).
- Tiré du site Web de Laurent Penet
- Nietzsche, La généalogie de la morale, Folio p. 85
- Plusieurs paragraphes de cette partie "évolution et réseau de régulation " se basent largement sur les chapitres 10 et 11 de l'ouvrage de Uri Alon, An introduction to system biology , Chapman & Hall.
- En réalité, il s'agit de l'allolactose.