Evolution (II)
"Anti-Darwin. Pour ce qui est de la fameuse lutte pour la vie, elle me semble jusqu'à présent plus souvent proclamée que prouvée. Elle peut avoir lieu mais c'est l'exception: le caractère le plus général de la vie, ce n'est nullement la pénurie, la famine, c'est plutôt la richesse, l'opulence et même l'absurde gaspillage."1
Préambule
Dans le premier chapitre sur l'évolution, j'ai essayé de rester relativement "positiviste" : le lecteur y a trouvé de nombreuses citations de publications scientifiques. Dans ce chapitre, il n'y aura pas de citations de publications. Nous allons aborder des aspects plus théoriques mais également plus polémiques, spéculatifs voire métaphysiques. La première partie traite des critiques/alternatives possibles à la théorie de l'évolution. Le socle de mon argumentation sera basé sur les travaux de Stuart Kauffman. La deuxième partie de ce chapitre reprend le débat sur les unités de sélection. J'essaie d'y décrire une vision plus personnelle.
Critiques et alternatives à la théorie de l'évolution
Quand on s'apprête à débattre/discuter/attaquer/critiquer la théorie de l'évolution, le risque est grand que votre "adversaire" (un scientifique) se braque et/ou se ferme comme une huitre. Il a souvent l'impression que l'on s'apprête à aborder le débat classique qui existe entre créationnistes et évolutionnistes. Dans ce débat, les échafaudages cognitifs qui amènent à défendre l'une ou l'autre de ces doctrines/théories sont si différents que les scientifiques entrevoient immédiatement que leurs arguments ne pourront pas convaincre. L'intérêt du débat devient alors uniquement politique.
Dans mon cas, je souhaite "discuter" de la théorie de l'évolution en utilisant, il me semble, le même échafaudage cognitif que les scientifiques. Mon but est donc bien de convaincre le lecteur scientifique à chercher à coté ou aux frontières du paradigme darwinien (et ne pas se contenter de chercher à l'intérieur). L'idée n'est pas de demontrer que la théorie de l'évolution est fausse mais plutôt de chercher si il est possible de l'englober dans quelque chose de plus large, de plus complet, de plus prédictif (comme la théorie de la gravitation de Newton a été englobée par la théorie de la relativité générale d'Einstein).
Karl Popper lui-même a mis en cause la théorie de l'évolution2 en arguant que celle-ci n'est pas falsifiable et relève donc plus de la métaphysique:
"I have come to the conclusion that Darwinism is not a testable scientific theory, but a metaphysical research program"
Puis il s'est finalement rétracté:
"I have changed my mind about the testability and logical status of the theory of natural selection; and I am glad to have an opportunity to make a recantation"
Ce qui gène avec la théorie de l'évolution c'est qu'elle semble pouvoir expliquer n'importe quelle fonction/structure ordonnée trouvée dans la nature. Or une théorie capable d'expliquer "tout et n'importe quoi" est-elle vraiment une théorie scientifique ? La réponse est oui car la théorie de l'évolution est une théorie explicative mais non prédictive. Prenons un exemple : l'apparition de la fonction "oeil" peut-être expliquée avec des concepts de mutations, de sélection et de contraintes mais si je prends une séquence ADN de 4 milliards de paire de bases faite que de A (adénine), je ne peux pas prédire la liste des mutations susceptibles de conduire à la fonction "oeil".
Je pense qu'il faut donc partir de ces deux mots "explicatif" et "prédictif" et se poser les questions suivantes:
- Y a-t-il des fonctions/structures ordonnées qui ne peuvent pas être expliquées par la théorie de l'évolution. Si oui lesquelles et pourquoi ? A l'inverse quelles sont les propriétés communes aux fonctions explicables par la théorie de l'évolution ?
- Nous verrons dans la suite que le paysage adaptatif sur lequel les organismes évoluent est un hypercube. Quelles sont les propriétés de cette hypercube, a-t-il un structure particulière dont la connaissance nous fournirait des outils pour devenir prédictif ?
Cette première partie sera divisée en 3 exemples issus de la théorie de Stuart Kauffman, un biologiste théoricien américain. Il a écrit un ouvrage The origin of order dans lequel il propose une alternative à l'idée que l'ordre que l'on observe dans les organismes vivants provient uniquement des effets de la sélection naturelle. Pour écrire cette partie, je me suis basé sur l'ouvrage At home in the universe qui représente une version simplifiée et très accessible de The origin of order. J'ai également utilisé l'ouvrage d'Hervé Zwirn Les systèmes complexes qui reprend en français la description du modèle NK. Mon texte est peut être une synthèse un petit peu abrupte : je renvoie donc le lecteur avide de clarté vers ces deux ouvrages.
Si vous avez l'occasion de lire l'ouvrage At home in the universe, vous vous rendrez compte que ce dernier est truffé de conditionnels. L'auteur se sent obligé de mettre en doute ses propres idées et parle de sa théorie comme d'une métaphore "more of a metaphor than the start of a real theory" 3 . Cette attitude humble reflète les doutes de l'auteur mais également, il me semble, les oeufs sur lesquels il a marché en proposant sa théorie. Autrement dit "ses excuses" reflètent l'attitude dogmatique des scientifiques vis-à-vis d'une théorie de l'évolution devenue "intouchable".
Le métabolisme primitif
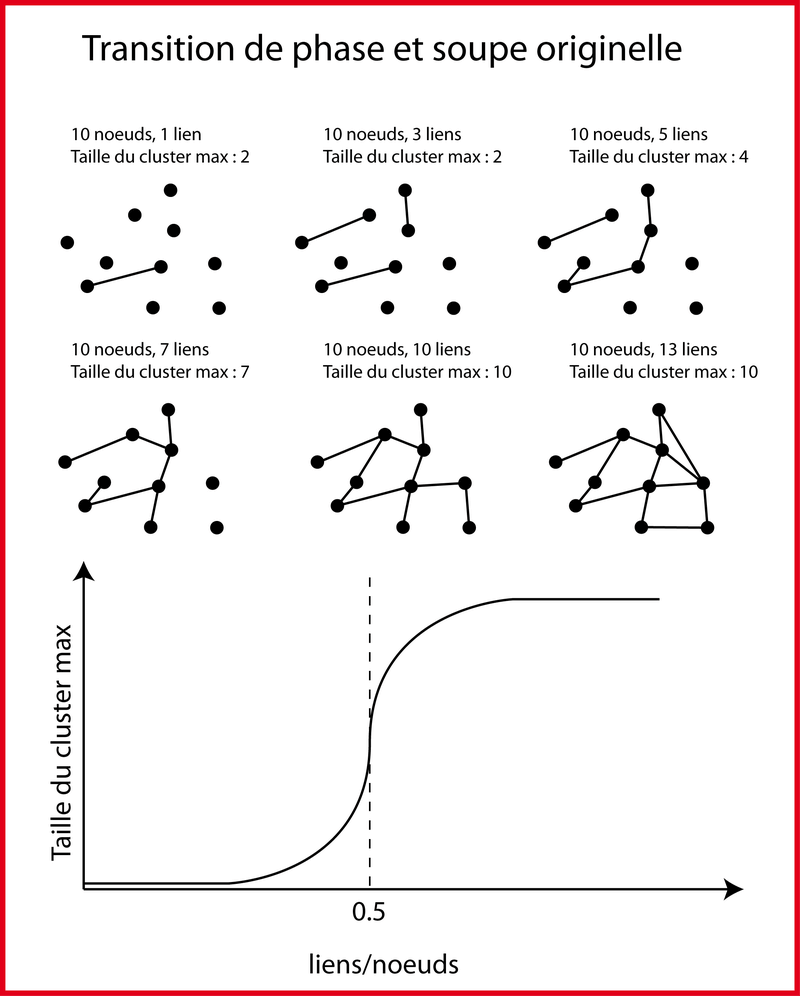
Commençons par un petit exemple simple : une sorte de cas d'école. Imaginons un graphe (un réseau) aléatoire constitué de noeuds et de liens connectés ensemble aléatoirement (figure 1). Au départ, on considère 1000 noeuds non connectés (l'exemple dans la figure 1 en contient 10). Puis on choisit 2 noeuds au hasard que l'on connecte ensemble avec un lien et on cherche le cluster (noeuds connectés ensemble) le plus grand. A ce stade, le plus grand cluster contient 2 noeuds. Puis on itère ce processus : on choisit 2 nouveaux noeuds au hasard qu'on réunit par un lien et on cherche le cluster le plus grand et ainsi de suite jusqu'à ce que ce cluster contiennent l'ensemble des 1000 noeuds. Au début, lorsque que l'on augmente le nombre de noeuds, la taille du cluster stagne et reste faible. Puis lorsque le ratio liens/noeuds dépasse ½, comme par magie, un cluster géant se forme soudainement (visible pour 10 noeuds mais plus évident avec 1000) car la plupart des petits clusters déjà formés se connectent ensemble d'un coup. On parle de transition de phase. Selon Stuart Kauffman, ce type de transition pourrait avoir été à l'origine de l'apparition de la vie. Dans notre réseau, les noeuds seraient des molécules (à pouvoir catalytique) et les liens des réactions chimiques. Une fois que le ratio réactions/molécules dépasse 0.5, un vaste réseau de réactions catalysées émerge/cristallise soudainement créant une sorte d'ensemble auto-catalytique capable de se maintenir par lui-même. Vous voyez que l'ordre présent dans ce métabolisme primitif n'est pas lié à la présence d'évolution ou de sélection mais plutôt aux propriétés des réseaux aléatoires.
"The intuition I want you to take away from this toy problem is simple: as the ratio of threads to buttons increases, suddenly so many buttons are connected that a vast web of buttons forms in the system. This giant component is not mysterious; its emergence is the natural, expected property of a random graph. The analogue in the origin-of-life theory will be that when a large enough number of reactions are catalyzed in a chemical reaction system, a vast web of catalyzed reactions will suddenly crystallize. Such a web, it turns out, is almost certainly auto-catalytic-almost certainly self-sustaining, alive." 4
Le réseau de régulation génique
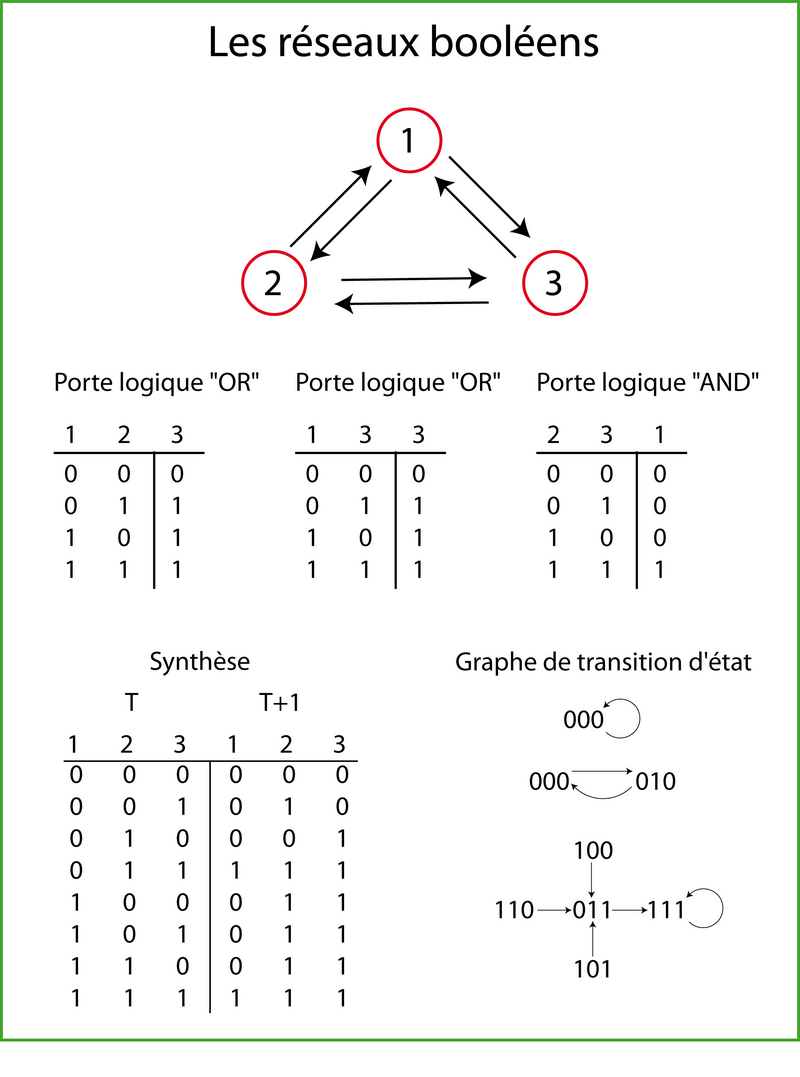
Stuart Kauffman est sans doute l'un des premiers scientifiques à avoir proposé l'utilisation de réseaux booléens pour décrire la dynamique des réseaux de régulation génique. Dans un réseau booléen, les noeuds (gènes) sont reliés par des flèches (les interactions géniques). Chaque gène est soit allumé (1) soit éteint (0). Un réseau booléen opère en temps discret (c'est un type d'automate cellulaire). La valeur des gènes à un temps t représente l'état du réseau. A t+1, chaque gène adopte un nouvel état en fonction des flèches du réseau. Au fur et à mesure que le temps passe, et en fonction des états initiaux des gènes, la séquence des états converge vers un cycle attracteur. En connaissant l'ensemble des états et trajectoires que draine un attracteur, on peut tracer le bassin d'attraction correspondant.
Prenons l'exemple d'un réseau booléen à trois gènes (figure 2). Dans notre exemple,
- l'état du gène 3 au temps t+1 est donné par l'état des gènes 1 et 2 au temps t selon la fonction OR.
- l'état du gène 2 au temps t+1 est donné par l'état des gènes 1 et 3 au temps t selon la fonction OR.
- l'état du gène 1 au temps t+1 est donné par l'état des gènes 2 et 3 au temps t selon la fonction AND.
L'ensemble des cas possibles est présenté dans le tableau de la figure 2. Si l'état initial du réseau est 000, il restera dans cette configuration. Si l'état initial est 010 ou 001, il oscillera indéfiniment entre ces deux configurations. Dans tous les autre cas (011, 100, 101, 110,111), le système finira par se stabiliser sur 111. Dans nos exemples, 000 et 111 sont des cycles attracteurs de longueur 1 : une fois que le système atteint cet état, il y reste. 010/000 forme un cycle attracteur de longueur 2. On peut imaginer des cycles attracteurs passant successivement par tous les états possibles du système. Pour un réseau de 3 gènes, il y a 23 états possibles. Pour un réseau de n gènes, il y a 2n états possibles : Si on considère le génome humain (~30 000 gènes), il y a 230000 configurations possibles : un nombre qui défie l'entendement (et l'ordinateur !).
Dans son modèle NK, Stuart Kauffman fixe le nombre N de gènes (noeuds) et fait varier le nombre moyen d'interactions (liens) K régissant l'état de chaque gène. C'est un réseau booléen aléatoire : les portes logiques ainsi que les flèches qui unissent les gènes sont choisis aléatoirement. Puis Stuart Kauffman se focalise sur les deux questions suivantes :
- Combien y-a-t-ils de cycles attracteurs ?
- Quelle est la longueur des cycles attracteurs ?
Lorsque K est égal à N (voir l'exemple de la figure 3), chaque gène dépend de l'ensemble
des autres gènes. Le réseau est totalement connecté. La longueur moyenne des cycles attracteurs est de
l'ordre de la racine carré du nombre de configurations. Le nombre de cycles attracteurs est de l'ordre
de N/e (où e est la base des logarithmes népériens et vaut environ 2.718). Si on prend N= 30 000 gènes
(soit le nombre de gènes du génome humain), le nombre de cycle attracteur est de l'ordre de 10 000 et la
longueur moyenne des cycles attracteurs est de l'ordre de 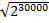 c'est-à-dire un nombre
astronomique. Cette longueur est telle qu'il est impossible de voir autre chose qu'un comportement
chaotique. Le système parcourt chacun des
c'est-à-dire un nombre
astronomique. Cette longueur est telle qu'il est impossible de voir autre chose qu'un comportement
chaotique. Le système parcourt chacun des 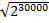 états
avant de revenir au premier état. De
plus, ce type de réseau est très sensible aux conditions initiales : le simple fait de changer un 1 ou
un 0 le fait basculer vers un autre cycle attracteur.
états
avant de revenir au premier état. De
plus, ce type de réseau est très sensible aux conditions initiales : le simple fait de changer un 1 ou
un 0 le fait basculer vers un autre cycle attracteur.
En revanche, lorsque k=2 soit deux interactions en moyenne par gène, la longueur des cycles devient égale non plus à la racine carré du nombre de configurations (2N) mais à la racine carré du nombre de gènes (N). De plus, le nombre d'attracteurs est également de l'ordre de la racine carré du nombre de gènes (N). Autrement dit, le système reste confiné dans une portion minuscule de son espace des configurations. Une transition de phase dans le comportement du réseau se produit et l'ordre apparait. De plus, le réseau est très stable : en changeant un 1 ou un 0, le système reste le plus souvent dans le même bassin d'attraction. Seule quelques rares "mutations" permettent de "switcher" sur un autre attracteur.
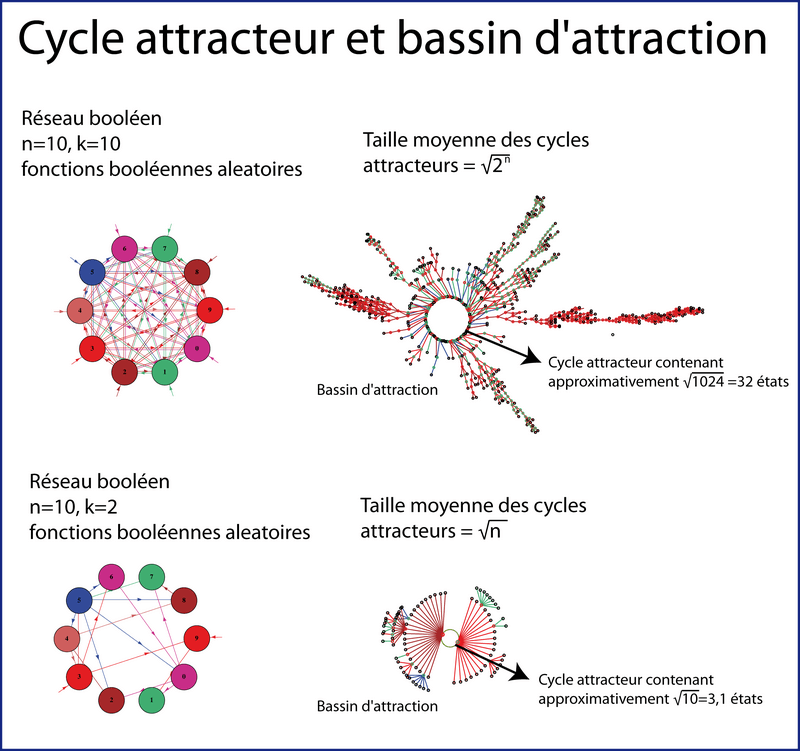
Ainsi lorsque K est inferieur à 2, le comportement du système est ordonné et fiable alors que lorsque K est supérieur à 3, le comportement du système est instable et chaotique. Selon Stuart Kauffman, l'évolution (la vie) est contrainte de se placer à la frontière entre l'ordre et le chaos c'est-à-dire au niveau de la transition de phase qui s'opère en "tunant" K.
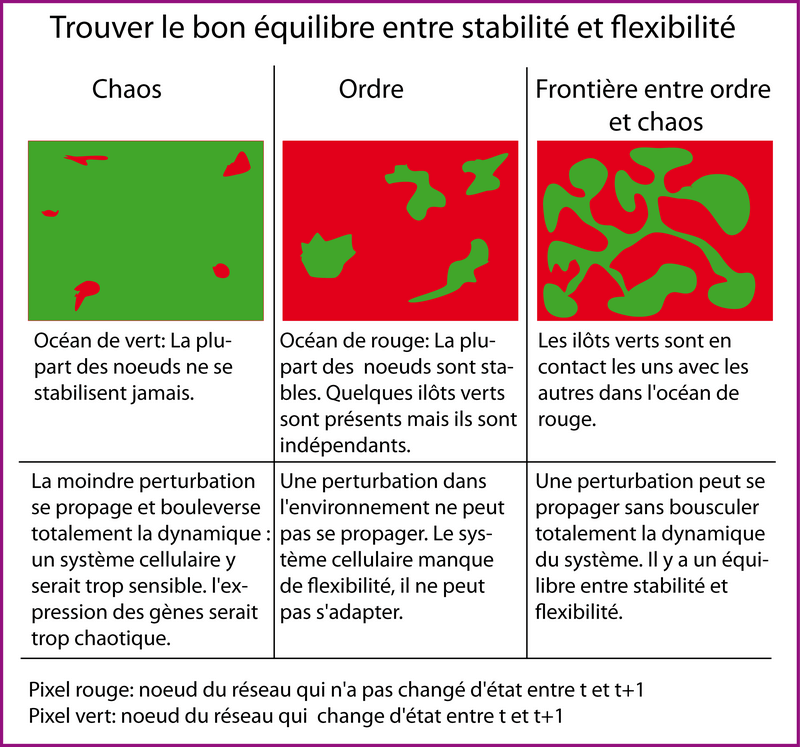
Pour illustrer cette idée, Stuart Kauffman nous propose une image en couleur (figure 4). Lors du passage de l'instant t à t+1, un noeud peut changer d'état. Colorons en rouge les noeuds du réseau qui n'ont pas changé d'état et en vert les autres. Quand K est grand (>3), le réseau est chaotique : on observera un océan de vert avec quelques îlots minuscules rouges : les noeuds ne se stabilisent jamais. Si on considère une petite perturbation dans l'environnement qui viendrait modifier l'expression d'un gène (changer son état de 0 à 1), cette perturbation se propagerait à l'ensemble du réseau en bouleversant totalement sa dynamique. La sensibilité du système est telle qu'une cellule ne pourrait jamais coordonner son comportement. Si on regarde maintenant un réseau très ordonné, au début tout est vert, puis au fur et à mesure que l'on converge vers un cycle, le rouge s'étend et à la fin la plupart des noeuds sont rouges. Seuls quelques îlots verts indépendants perdurent, ceux qui participent au cycle. Une perturbation dans ce système ne pourrait pas se propager car les îlots verts sont déconnectés les uns des autres. Le système est figé, la cellule ne peut pas modifier son comportement en réponse à un changement environnemental. Enfin, à la frontière entre l'ordre et le chaos, les îlots verts sont en contact ténu les uns avec les autres dans l'océan de rouge. La plupart des perturbations seront sans conséquence sur la cellule, mais certaines permettront de "switcher " d'un îlot vert à l'autre : la cellule se coordonne, s'adapte sans pour autant bousculer toute la dynamique du système. Pour Hervé Zwirn :
"Les systèmes adaptatifs fonctionneraient dans le régime ordonné qui se situe à la frontière de la transition entre ordre et chaos. Ils doivent en effet respecter un subtil équilibre entre stabilité et flexibilité"5
Stuart Kauffman avance plusieurs arguments biologiques en faveur de l'idée que les réseaux de régulation génique aient un K proche de 2.
- Premièrement, les gènes ont souvent un petit nombre "d'input" commandant leurs expressions: seuls quelques facteurs de transcription régulent l'activité d'un promoteur.
- Deuxièmement, on a vu que lorsque N=30 000 (le nombre de gènes présents dans le génome humain) et K=2, le nombre d'attracteurs est de l'ordre de la racine carré de N soit 173. Or dans un environnement donné, le nombre de type de cellules différenciées chez l'homme (équivalent des cycles attracteurs) est de 256, un nombre très proche de 173. Enfin, la longueur moyen d'un cycle attracteur est de 173 or si on considère qu'il faut 10 minutes pour activer ou réprimer un gène, le temps pour accomplir un cycle sera de l'ordre de 173x10 soit environ 30 heures ce qui est précisément la bonne échelle de temps pour le comportement d'une cellule humaine.
Ainsi pour Stuart Kauffman, l'ordre généré par un tel réseau est de l'ordre "gratuit" c'est-à-dire qui ne provient pas des cycles et de cycles de sélection naturelle. L'évolution est contrainte de "tuner" ses paramètres (par exemple K) pour être et rester à la frontière entre ordre et chaos. L'évolution est ici subordonnée à des lois relatives au fonctionnement du réseau.
"The reason complex systems exist on, or in the ordered regime near, the edge of chaos is because evolution takes them there.[...] perhaps natural selection then tunes their parameters, tweaking the dials for K and P, until they are in the ordered regime near this edge- the transitional region between order and chaos where complex behavior thrives. After all, systems capable of complex behavior have a decided survival advantage, and thus natural selection finds its role as the molder and shaper of the spontaneous order for free;" 6
Le réseau épistatique
Nous allons maintenant étudier un deuxième type de réseau présent dans les organismes vivants : Le réseau épistatique. Vous ne comprendrez que tardivement le lien avec le sujet de cette première partie : l'alternative à la théorie de l'évolution. En effet, il me faut tout d'abord expliciter la notion de paysage adaptatif (fitness landscape). Je précise tout de suite que je vais introduire une nuance importante par rapport aux travaux de Stuart Kauffman. Les paysages adaptatifs de ce dernier ont comme unité de base le gène or je pense qu'il est préférable d'utiliser le nucléotide.
La notion de paysage adaptatif
La chose la plus importante à garder à l'esprit, c'est que la notion de paysage adaptatif (terme inventé dans les années 40) est une image. Il faut donc imaginer un paysage en 3 dimensions, fait de plaines et de montagnes. L'axe Z représente le fitness. Les axes X et Y forment une grille de tous les génomes possibles d'une certaine taille. Les sommets des montagnes représentent des hautes valeurs de fitness, c'est-à-dire de fortes capacités à se reproduire. Il faut se représenter l'évolution comme la lutte d'une population d'organismes, via les mutations et la sélection pour grimper au sommet de la plus haute montagne : le maximum global. Imaginons un génotype donné dont la valeur de fitness le place en bas d'une montagne dans le paysage adaptatif. Comment se passe la marche adaptative ? une mutation apparait au hasard dans le génome. Puis le fitness de ce nouveau génome est évalué. S'il est supérieur, on monte le long de la montagne. S'il est inferieur, on ne fait rien.
Le problème, c'est que le vrai paysage adaptatif ne peut pas être représenté en 3 dimensions. L'axe Z ne pose pas de problème : c'est le fitness. Mais au dessus d'un génome de 2 nucléotides, les axes X et Y ne suffisent plus à représenter toutes les possibilités de génomes possibles. Pour un génome de 2 nucléotides, pas de problème. Il y a 16 possibilités : AA, AT, AC, AG, TA, TT, TC, TG, CA, CT, CC, CG, GA, GT, GC, GG. Hypothétiquement (en fait, on ne peut bien sur rien faire avec un génome de 2 nucléotides), je peux synthétiser ces fragments d'ADN, les transformer dans un châssis cellulaire et mesurer pour chaque combinaison le fitness associé. Puis je dresse le paysage adaptatif comme ci-dessous : le premier nucléotide est sur l'axe X (4 possibilités : A, T, C et G) et le deuxième nucléotide est sur l'axe Y (4 possibilités : A, T, C et G). Ce paysage adaptatif ressemble bien à un paysage montagneux (figure 5). Mais vous voyez que si je rajoute un nucléotide (génome de 3 nucléotides), j'ai maintenant un problème car je n'ai plus de dimensions à ma disposition puisque que X, Y et Z sont déjà utilisés. Au dessus de 2 nucléotides, nous sommes obligés de rajouter des dimensions qui ne sont pas représentables (par définition) en 3 dimensions. Pour un génome de n nucléotides, le paysage adaptatif correspondant possède 2n dimensions. Pour les matheux, c'est un hypercube de dimension 2n. Pour les biologistes, le paysage adaptatif d'E. coli nécessiterait "seulement" 8 millions de dimensions pour être représenté alors que notre esprit dit "stop" au delà de 3 dimensions.

Mais peut-on connaitre tout de même un peu l'allure de ce paysage adaptif même si la représentation tridimensionnelle n'est qu'une image ? Et surtout, peut-on savoir à quoi il ne ressemble pas ? La réponse est oui et c'est ce que nous allons voir maintenant.
Le paysage adaptatif sans corrélation
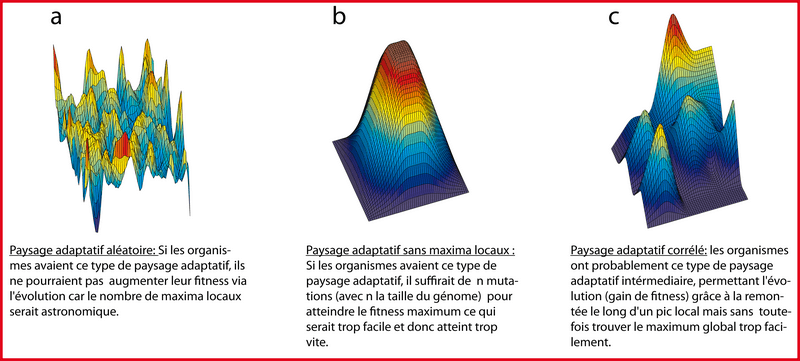
Dans un paysage adaptatif sans corrélation, chaque nucléotide du génome est strictement indépendant. Imaginons un génome de 100 nucléotides indépendants. Il y a 4100 génomes possibles différents. Puis admettons un génome de 100 paires de bases composé uniquement de A (donc 100 Adénines les unes à la suite des autres). Nous décidons que ce génome est celui qui a le meilleur fitness : l'optimum global. Notez que ce choix semble être particulier alors qu'il ne l'est pas : ce génome a exactement la même probabilité d'être le plus "fit /adapté" que les (4100-1) autres. Puis créons un génome aléatoire de 100 nucléotides. Il y aura approximativement 25% de chaque base A, T, C, G. Faisons évoluer ce génome à la recherche de l'optimum global c'est-à-dire le génome de 100 A. Comme les nucléotides sont indépendants les uns des autres, chaque A qui apparaitra dans le génome aléatoire apportera un gain de fitness et sera donc sélectionné. Ceci a pour conséquence qu'on ne s'intéresse pas aux mutations qui apparaissent dans les A car elles ne seront pas sélectionnées/fixées étant donné qu'elles diminuent le fitness. A l'inverse toute mutation qui apparait sur les bases T, C ou G du génome aléatoire a une chance sur 3 d'être un A. il faudra donc en moyenne à peine 3x100 mutations pour obtenir le génome optimal c'est-à-dire le génome le plus adapté composé que de A. Pour le génome d'E. coli de 4 millions de nucléotides, le nombre de combinaisons (mutations) à tester pour trouver l'optimum global serait de 12 millions. Ce chiffre peut paraître grand mais il est en réalité minuscule. Si les choses étaient aussi simples, les 4 milliards d'années d'évolution auraient largement suffit à trouver le génome optimal via 12 millions de mutations. Or E. coli avec son taux de croissance de 20 minutes ne possède pas un génome optimal. L'algorithme évolutif tourne encore pour améliorer ce fitness. Rien n'empêche de penser qu'il existe un génome hypothétique de 4 millions de paires de bases permettant à E. coli de se diviser toutes les 2 minutes. Aucun des génomes présents sur terre n'a atteint l'optimum global. Or, avec un paysage adaptatif sans corrélation, le maximum global est très facile à trouver car il ne nécessite qu'un nombre de mutation très faible, de l'ordre de grandeur de la taille du génome. Ce paysage adaptatif ressemble au Mont Fuji au Japon (figure 6 et figure 7b).

Le paysage adaptatif corrélé
Une conclusion : les paysages adaptatifs des organismes ne ressemblent pas au mont Fuji. La seule solution possible pour empêcher cela consiste à supprimer la clause d'indépendance des nucléotides entre eux. Les nucléotides d'un génome ne sont pas indépendants, ils sont reliés par des interactions virtuelles. Stuart Kaufmann parle d'épistasie, c'est-à-dire les interactions virtuelles qui existent entre deux ou plusieurs gènes. Par exemple, un gène A qui permet d'importer le glucose dans une cellule a une relation d'épistasie avec un gène B qui permet d'utiliser ce glucose comme énergie. Le gain de fitness apporté à la cellule n'apparait que si A et B sont présents. Le gène A sans le gène B (et réciproquement) ne sert à rien. Un fil invisible unit ces deux gènes. A l'échelle des 4000 gènes d'E. coli, il existe donc un réseau complexe d'interactions épistatiques qui n'a rien à voir avec le réseau de régulation génique. Vous comprenez peut être maintenant une des raisons qui m'a poussé à faire, dans ma thèse, un détour par l'évolution : pour traiter correctement la question des réseaux chez E. coli, je ne pouvais pas ignorer les réseaux épistatiques. Il est facile de se représenter le réseau épistatique reliant des gènes entre eux. Cependant, je le répète, l'unité de base est le nucléotide et il est donc beaucoup plus juste et rigoureux de parler d'épistasie inter-nucléotidique. Je traite de ce sujet plus en profondeur dans mon chapitre sur l'information et j'y renvoie le lecteur. L'important est de comprendre que les nucléotides "se parlent" entre eux. Donnons un exemple: dans un codon stop TAA d'un gène codant pour une protéine essentielle, le T a une relation d'épistasie avec les deux A. Si, par exemple, le dernier A était un C, le codon deviendrait TAC, ce qui n'est pas un codon stop. Par conséquent, la protéine essentielle deviendrait trop longue, potentiellement non fonctionnelle et donc le fitness chuterait que le T soit présent ou non. Ainsi le T n'apporte le fitness à la bactérie que si les deux A son présents (ou s'ils forment un autre codon stop : TGA et TAG). L'absence d'indépendance permet d'éviter d'avoir un paysage adaptatif en forme de mont Fuji ce qui serait horrible car nous serions déjà des être parfaits depuis longtemps... 😀
Pour introduire cette communication inter-nucléotidique, je vais vous parler du modèle NK de Stuart Kauffman relatif au réseau épistatique. Un génome peut être composé de N gènes et chacun de ces N gènes peut avoir K interactions épistatiques avec les autres gènes. Dans notre cas, c'est presque exactement la même chose mais on "switch" en mode "nucléotide" : un génome peut être composé de N nucléotides et chacun de ces N nucléotides peut avoir K interactions épistatiques avec les autres nucléotides. Dans le modèle NK, un nucléotide peut avoir au minimum K=0 interaction avec les N-1 autres nucléotides. Dans ce cas, les nucléotides sont indépendants les uns des autres et le paysage adaptatif a une forme de Mont Fuji. Dans le modèle NK, un nucléotide peut avoir au maximum K=N-1 interactions avec les autres nucléotides. Dans ce cas, chaque nucléotide exerce une influence sur tous les autres. Le paysage adaptatif ressemble à un paysage totalement aléatoire (figure 7a). Le nombre de maxima locaux devient très vite astronomique quand N croît ce qui signifie qu'une population d'organismes ne peut évoluer dans se paysage. Elle tombera sans cesse sur des maxima locaux. La marche adaptative (l'algorithme évolutif) est totalement inutile car la recherche revient à tester des génotypes au hasard. Une population ne pourrait fouiller qu'une quantité infime du paysage. La seule solution pour trouver le maximum global consiste à essayer une par une toutes les possibilités soit, pour un génome de 100 nucléotides, 4100 soit plus que le nombre d'atomes dans l'univers connu (1080). Le temps de Planck est la plus petite unité de temps mesurable dans le cadre de nos théories. Il est de 5x10-44 secondes. L'âge de l'univers est estimé à environ 14 milliards d'années, soit 4x1017 secondes. Si on mesurait le fitness d'un génotype à chaque temps de Planck, on aurait pu tester, depuis la création de l'univers, seulement 20x1061 génomes soit encore loin de 4100 requis pour déterminer le génome possédant le meilleur fitness.
Cliquer pour voir le code Matlab
% petit script pour génerer des paysages adaptatifs (fitness landscape)
figure('Color','w')
for i=1:3
Handle(i)=subplot(1,3,i);
axis(Handle(i),'off');
hold(Handle(i));
view(-42,26);
end
% plot 1
[x,y]=meshgrid([0:1:5]);
A=[0 0 0 0 0 0; 0 1 2 2 1 0; 0 3 4 4 3 0; 0 3 4 4 3 0;...
0 1 2 2 1 0; 0 0 0 0 0 0 ];
[XI,YI] = meshgrid(0:.1:5);
ZI = interp2(x,y,A,XI,YI);
x = {linspace(0,6,51),linspace(0,6,51)};
[xx,yy] = ndgrid(x{1},x{2}); y = ZI;
[smooth,p] = csaps(x,y,[],x);
smoother = csaps(x,y,.996,x);
surf(Handle(1),x{1},x{2},smoother.'), axis off
% plot 2
[x,y]=meshgrid([0:1:5]);
A=rand(6,6)*5;
[XI,YI] = meshgrid(0:.1:5);
ZI=rand(51,51)*5;
x = {linspace(0,6,51),linspace(0,6,51)};
[xx,yy] = ndgrid(x{1},x{2}); y = ZI;
[smooth,p] = csaps(x,y,[],x);
smoother = csaps(x,y,.996,x);
surf(Handle(2),x{1},x{2},smoother.'), axis off
% plot 3
[x,y]=meshgrid([0:1:5]);
A=[0 1 0 1 0 0; 0 4 0 3 1 1; 0 0 0 0 1 1; 0 0 1 3 1 5;...
0 0 0 2 2 3; 0 0 3 0 2 3 ];
[XI,YI] = meshgrid(0:.1:5);
ZI = interp2(x,y,A,XI,YI);
x = {linspace(0,6,51),linspace(0,6,51)};
[xx,yy] = ndgrid(x{1},x{2}); y = ZI;
[smooth,p] = csaps(x,y,[],x);
smoother = csaps(x,y,.996,x);
surf(Handle(3),x{1},x{2},smoother.'), axis off
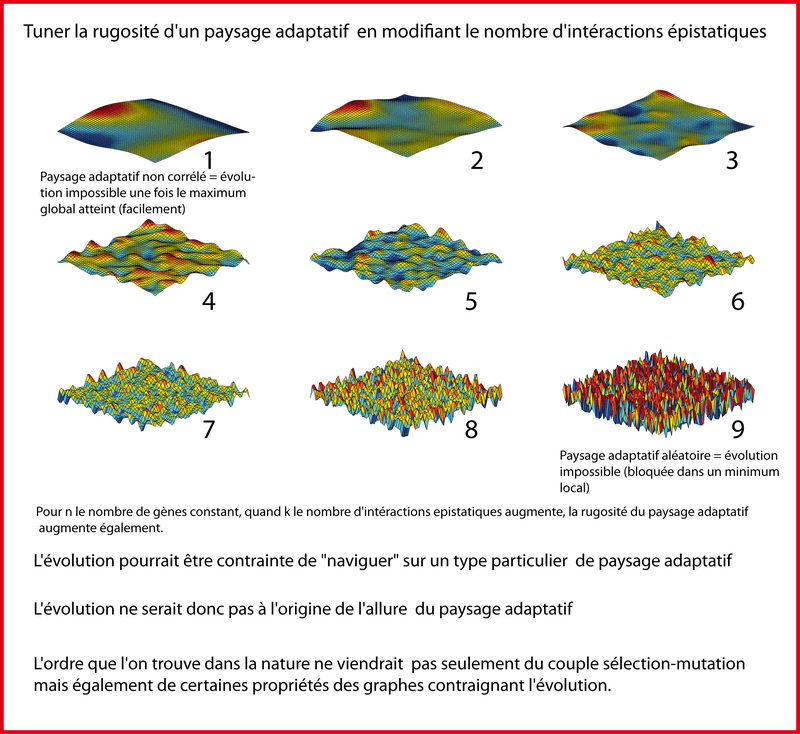
Les choses ne se passent donc pas de cette manière dans la nature. Les populations d'organismes arrivent à utiliser la marche adaptative ce qui signifie que le paysage adaptatif ne peut pas être entièrement aléatoire. En fait, en faisant varier K, on contrôle la rugosité du paysage adaptatif (figure 8). Dans la nature, K doit être judicieusement choisi entre 0 et N-1 de manière à former un paysage adaptif "navigable"8 comme celui de la figure 7c. La rugosité du paysage adaptatif détermine son "evolvabilité". Mais qui est à l'origine de cette evolvabilité? est-ce-que c'est l'évolution elle-même ? L'évolution peut-elle contrôler/tuner/faire évoluer l'evolvabilité? Ou est-ce-que l'evolvabilité est encore une fois une caractéristique intrinsèque du réseau épistatique à laquelle est subordonnée l'évolution?
Cliquer pour voir le code Matlab
% petit script pour tuner "faussement" la rugosité d'un paysage adaptatif.
% en effet, ce script ne se base evidemment pas sur le modèle NK car on ne
% peut pas representer un paysage adaptatif de plus de 2 nucléotides en 3
% dimensions.
figure('Color','w')
for i=1:9
Handle(i)=subplot(3,3,i);
axis(Handle(i),'off')
hold(Handle(i));
view(44,60);
end
for i=1:9
Fit=[ 0.1 0.7 0.9 0.99 0.995 0.999 0.9995 0.9999 1];
[x,y]=meshgrid([0:1:5]);
[XI,YI] = meshgrid(0:.1:5);
ZI=rand(51,51)*5;
x = {linspace(0,6,51),linspace(0,6,51)};
[xx,yy] = ndgrid(x{1},x{2}); y = ZI;
[smooth,p] = csaps(x,y,[],x);
smoother = csaps(x,y,Fit(i),x);
surf(Handle(i),x{1},x{2},smoother.'), axis off
end
"Those who believe that natural selection is the sole source of biological order must assume that selection itself achieved organisms with the proper level of epistasis, the right value of K. But is selection powerful enough to craft the structure of fitness landscapes? Or are there limits to the power of selection? If selection is too weak to ensure evolvability, how is such evolvability achieved and maintained? Could self-organization play a role? Here is a profound problem that must reshape our thinking." 9
"However, the very limits on selection we have discussed must raise questions about whether selection itself can achieve and sustain the kinds of organisms that adapt on the kinds of landscapes where selection works well. It is by no means obvious that selection can, of its own accord, achieve and sustain evolvability. Were cells and organisms not inherently the kinds of entities such that selection could work, how could selection gain a foothold? After all, how could evolution itself bring evolvability into existence, pulling itself up by its own bootstraps? And so we return to a tantalizing possibility: that self-organization is a prerequisite for evolvability, that it generates the kinds of structures that can benefit from natural selection."10
Les unités de sélection
Je vais maintenant passer à la deuxième partie de ce chapitre. Pour l'aborder, il faut avoir lu la partie précédente sur les paysages adaptatifs.
Le débat sur les unités de sélection cherche à mettre en évidence sur quelle entité opère la sélection naturelle. On a pensé pendant longtemps que celle-ci s'opérait sur l'organisme jusqu'à ce que Richard Dawkins propose que la sélection naturelle s'opère sur les gènes eux-mêmes. Ces derniers seraient égoïstes c'est-à-dire qu'ils favoriseraient leur copie parfois même au détriment de la survie de l'organisme qui les contient. Les opposants à l'idée que le gène est la seule unité de sélection défendent une vision plus hiérarchique. Selon eux, la sélection naturelle peut s'opérer à différents niveaux (gènes, génomes, cellules, organismes, espèces, populations, écosystèmes...). Thomas Pradeu11 note qu'une des clarifications les plus intéressantes provient de David Hull.
"La clarification qui fut probablement la plus utile est due à David Hull. Ce dernier propose de faire la distinction entre deux entités biologiques impliquées dans le processus évolutionnaire: le réplicateur, qui désigne "une entité qui transmet sa structure largement intacte dans des réplications successives" (i.e. une entité qui est fidèlement copiée), et l'interacteur, qui désigne "une entité qui interagit comme un tout cohésif avec son environnement d'une manière telle que cette interaction est la cause du caractère différentiel de la réplication" (i.e. une entité sur laquelle la sélection naturelle agit directement) [...]. Hull montre qu'il est clair que les meilleurs réplicateurs, dans l'état actuel de nos connaissances, sont les gènes (ce qui ne veut pas dire que ce sont les seuls) et donc que le véritable débat sur les "unités de sélection" concerne en réalité les seuls interacteurs. Une fois le débat clairement situé à ce niveau, la réponse de Dawkins convainc peu. [...] La réponse dominante au problème clarifié par Hull est [...] qu'il existe une hiérarchie d'interacteurs, dont le niveau le plus clairement établi est celui de l'organisme, le gène pouvant être, mais seulement parfois, un interacteur. L'organisme est en effet probablement le meilleur exemple d'interacteur, car c'est sur les traits phénotypiques de l'organisme que s'exerce principalement l'action de la sélection naturelle bien que ce dernier insiste tout autant sur l'idée que l'organisme n'est pas le seul interacteur. Dawkins a en partie reconnu ce point en développant sa thèse du "phénotype étendu". Cependant, pour Dawkins, la véritable entité sur laquelle s'exerce la sélection naturelle est, non pas l'organisme comme tel, mais l'ensemble des traits phénotypiques sur lesquels les gènes exercent leur influence, c'est-à-dire précisément le "phénotype étendu", qui peut aller bien au-delà des frontières de l'organisme. Par exemple, dans le cas d'un parasite, le système nerveux de l'organisme parasité peut faire partie du phénotype étendu du parasite." 1
j'aimerais maintenant apporter une vision plus personnelle à cette question des unités de sélection. Je n'ai pas étudié ce débat dans le détail mais il me semble, au regard du texte ci-dessus, que le consensus actuel fait la distinction entre le replicateur et l'interacteur. Je suis en parfait accord avec cette dichotomie et je vais maintenant tenter de préciser (avec le plus de modestie possible) comment je me représente le replicateur et l'interacteur. La notion centrale que je vais proposer est le concept d'interaction à la fois inter-nucléotidique et infra-nucléotidique. Certains des paragraphes qui suivent risquent de mettre à rude épreuve les capacités d'abstraction du lecteur. Je reconnais également que la clarté de mon texte n'est peut-être pas toujours optimale cependant, à défaut de garantir la vérité, je garantie l'existence d'un sens.
J'évacue tout de suite le problème du replicateur en précisant, bien que cela soit évident et je ne pense pas qu'il y ait matière à débat sur ce point, que l'unité de sélection du réplicateur n'est pas le gène comme noté dans le texte ci-dessus mais plutôt le nucléotide répliqué. En effet, le nucléotide est l'unité de base de la séquence ADN et correspond à 2 bits car il y a 4 possibilités : A, T, C et G (cela pourrait être codé avec 2 bits : 00, 01, 10, 11).
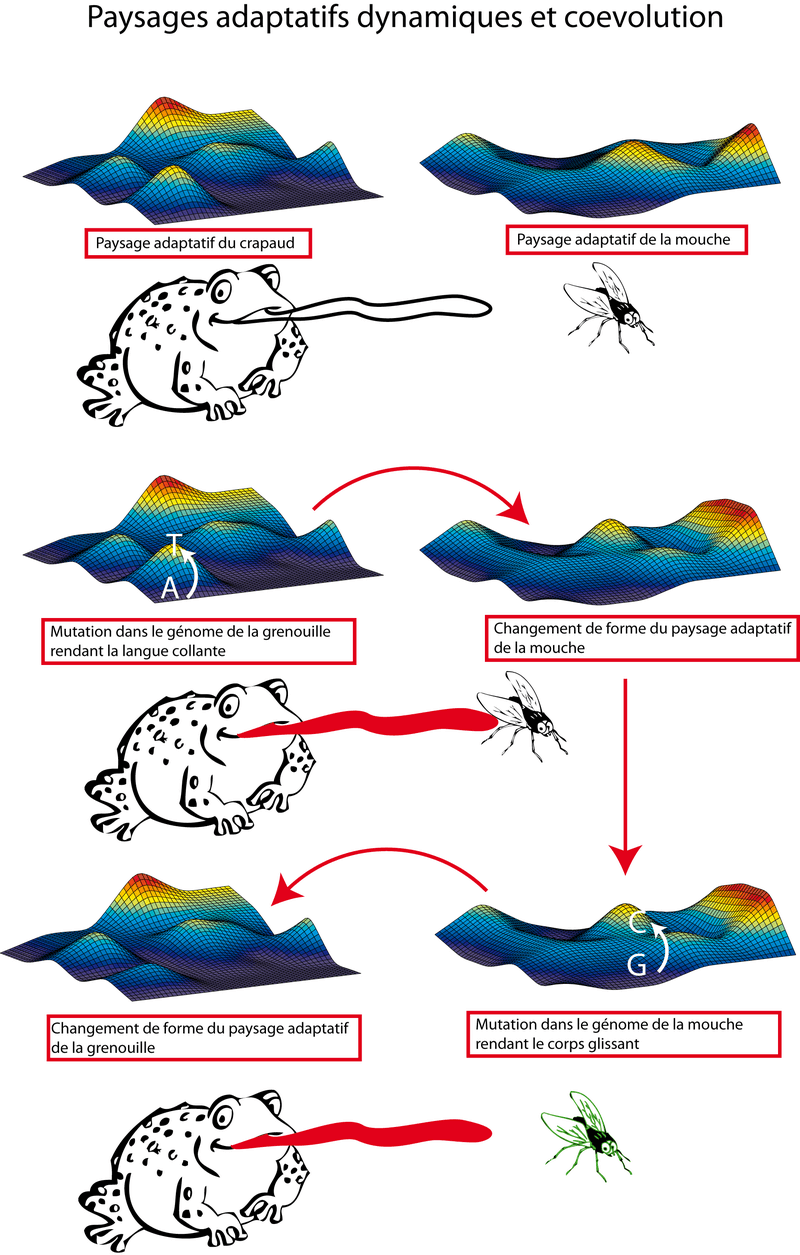
La coévolution
Dans la partie précédente, nous avons vu que les nucléotides sont connectés entre eux par un réseau épistatique. Mais ces interactions épistatiques inter-nucléotides ne sont pas limitées à l'organisme. De telles interactions peuvent exister entre des organismes différents. Prenons l'exemple connu (et que j'emprunte encore à Stuart Kauffman) du crapaud et de la mouche (Figure 9). Un crapaud a un certain génome qui forme un paysage adaptatif. Et il en va de même pour la mouche. Imaginons maintenant qu'une mutation apparaisse dans le génome du crapaud : par exemple un A devient un T. Cette mutation confère de meilleures propriétés collantes à la langue des crapauds. Le crapaud qui a la mutation attrape statistiquement les mouches plus souvent. Il a un meilleur fitness et transmet donc mieux son patrimoine génétique. Petit à petit, la population de crapauds avec la mutation grossit ce qui introduit des contraintes sur la population de mouches. Le paysage adaptatif des mouches se déforme, il y a une diminution du fitness de la population des mouches à cause de la mutation chez les crapauds. Des mouches mutantes (par exemple un G qui devient un C) avec un corps glissant sont sélectionnées puis deviennent majoritaires. Cet événement change le paysage adaptatif des crapauds en diminuant le gain de fitness associé à la mutation A->T. Ces phénomènes de coévolution ont parfois des conséquences étranges comme le "Red Queen Effect" : les organismes sont constamment obligés de fixer de nouvelles mutations pour maintenir le fitness constant. C'est exactement ce qui c'est passé ici avec les crapauds et les mouches : au final, une fois les deux mutations apparues, aucune des deux espèces n'a obtenu un gain de fitness.
Mais ce qui m'intéresse surtout, c'est que le A de départ des crapauds possède une relation virtuelle épistatique avec le G de départ des mouches. Cette relation épistatique virtuelle est, il me semble, un objet mathématique. Comme je ne sais pas trop de quel objet il s'agit, je propose en attendant mieux, qu'il s'agisse d'une fonction (voir chapitre sur l'information). Cette fonction decrirait l'interaction d'un nucléotide avec un ou plusieurs autres et prendrait un certain nombre de paramètres en argument. L'interaction virtuelle épistatique entre le A et le T a son pendant physico-chimique c'est-à-dire le contact physique entre les atomes de langues et les atomes de mouches. Ma conjecture est la suivante : toute interaction nucléotidique épistatique (décrite par une fonction difficile à conceptualiser) possède, à un moment ou à un autre, une réalité physique.
La difficulté réside dans le fait qu'il faut parfois passer par le milieu pour détecter l'expression d'interaction inter-nucléotidique. Prenons un autre exemple : les nucléotides du génome des arbres génèrent indirectement de l'oxygène qui compose notre atmosphère. Cet oxygène, nous devons le respirer pour vivre. Il se fixe physiquement à l'hémoglobine de nos érythrocytes (globule rouge) et permet la respiration. Il y a interaction virtuelle entre les nucléotides des arbres et nos nucléotides.
Ce que je décris a des liens évidents avec le concept de phénotype étendu de Richard Dawkins. Ce dernier argumente le fait que nous, biologistes, restreignons de manière arbitraire l'idée de phénotype en nous limitant à l'expression phénotypique des gènes d'un organisme sur son propre corps. Par exemple, selon Richard Dawkins, le nid d'un oiseau ou les monticules de terre des termites sont des phénotypes " étendus".

Cependant, les choses que je présente sont différentes. Je le répète : je passe d'une vue gène-centrée à une vue nucléotide-centrée. Car la notion de gène fait ressortir, selon moi, certains aspects anthropocentriques de la nature humaine. Nous, humains, " trouvons facile" de manipuler, de séparer des entités physiques distinctes. Ce marteau est différent de ce stylo. Cette enzyme A n'est pas cette enzyme B. Notre manière de comprendre une cellule consiste d'abord à comprendre ses entités et ce qui caractérise globalement une entité c'est le fait que ses atomes sont unis par des liaisons covalentes. Avec le nucléotide, c'est différent : la notion d'entité (et de liaison covalente) disparait. On baigne enfin dans un régime informatif strict car la correspondance nucléotide/bit est totale. En supprimant le gène, on supprime l'anthropocentrisme "entité". Ainsi si le concept "phénotype étendu" casse la barrière mentale de l'organisme (les gènes de l'oiseau génèrent un phénotype : le nid). Les interactions épistatiques inter-nucléotidiques inter-organismes permettent d'aller plus loin en s'affranchissant, non seulement de l'anthropocentrisme "organisme" mais aussi de l'anthropocentrisme "entité". Non : le gène ne représente pas une unité de sélection ni l'organisme d'ailleurs. L'unité de sélection de type "réplicateur" c'est le nucléotide. L'unité de sélection de type "interacteur" c'est une fonction décrivant une interaction entre nucléotides ayant une réalité physique que nous pouvons percevoir à différents niveaux (moléculaire, cellulaire, organisme, écosystèmes). La présence de la réalité physique, lors de l'exécution du programme génétique, permet la sélection naturelle. C'est donc cette fonction (ou une partie de cette fonction) qui est sélectionnée, qui est égoïste. En se débarrassant de "l'entité", on peut alors concevoir l'idée que cette fonction est un concept de niveau encore plus bas que le nucléotide car ce dernier est une unité physique de base "grossière" chargé d'encoder et de compresser l'information. Cette fonction est de plus "bas niveau" non pas sur le plan physico-chimique (en dessous du nucléotide, il y a les atomes et cela ne nous intéresse pas) : elle est de plus bas niveau sur le plan informatif. Dans un nucléotide, on peut compresser une grande quantité d'information, une sorte de pack d'interactions. Une seule de ces interactions est peut-être primordiale, sélectionnée, égoïste et les autres voyageant avec elle de manière opportuniste. L'unité de sélection de type "interacteur" est une fonction représentant une interaction inter-nucléotidique parmi un pack d'interactions. Le tout est bien caché derrière/dans l'unité de type "réplicateur" : le nucléotide.
A partir de là, il faut se pencher à nouveau sur le débat "réductionnisme/holisme". Pour faire simple, l'holisme affirme que la nature crée des ensembles qui sont plus que la somme de leurs parties. A l'inverse, le réductionnisme affirme que, pour comprendre un système, il faut le décomposer en éléments simples et donc que l'on peut expliquer le tout à l'aide des parties.
On comprend bien qu'il est difficile d'expliquer le fonctionnement d'un écosystème sans décrire le fonctionnement des organismes qui le composent. De même, on peut difficilement expliquer le fonctionnement d'un organisme sans décrire le fonctionnement des cellules. Et ainsi de suite: Ecosystème---organisme---cellule---molecule. Et le problème apparaît clairement lorsqu'apparait le "dernier" niveau : les atomes (j'exclue les particules élémentaires pour faire simple) car le retour dans le sens inverse semble alors impossible. En effet, une soupe d'atomes C, H, O, N, P, S, dont on connait parfaitement les propriétés, ne semble pas nous permettre de remonter à l'organisme ou à l'écosystème.
Dans ce débat, il me semble que le problème vient de la dimension "entité-centrée" alors qu'il faut réfléchir de manière "information-centrée". Avec cette manière de penser, il y a une première dichotomie qui n'est pas explicable en l'état actuel des connaissances:
- Un monde sans information (ou très pauvre en information) avec une soupe d'atomes et de molécules simples (par exemple : ce qu'on trouve sur une planète a priori sans vie comme la planète Mars).
- Un monde avec information comme la biosphère sur la planète Terre. Dans ce monde avec information, on peut trouver différent niveaux à envisager plutôt sous forme de boucle :
- L'unité de base du réplicateur : le nucléotide répliquée
- La séquence informative proprement dite (l'ADN par exemple)
- L'exécution du programme (le programme c'est la séquence ADN). Cela nécessite l'ADN avec un milieu (ce dernier incluant un châssis cellulaire, les lois physiques, le temps...) capable d'interpréter le programme.
- Mais en réalité, en dessous de l'unité de base du réplicateur (le nucléotide répliquée), on trouve l'interaction infra-nucléotidique mais celle-ci n'a de sens que lors de l'exécution du programme ADN ce qui permet de boucler la boucle (Figure 11).
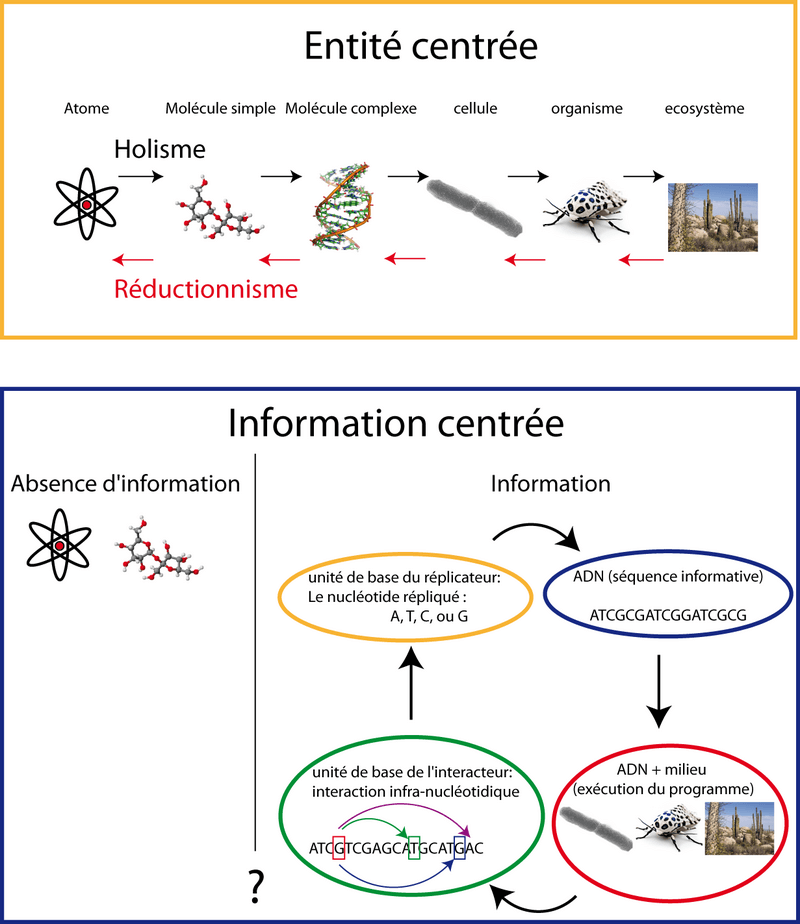
Avec cette vision, la question consistant à savoir si les atomes sont suffisants pour tout expliquer ou si l'émergence est une condition nécessaire disparait. La seule chose qui est nécessaire, pour expliquer tous les niveaux, c'est l'information. Et pour avoir de l'information, il faut, je le crois, disposer de séquences informatives un peu particulières. Mais je laisse mon lecteur se diriger vers le chapitre sur la vie s'il veut en savoir plus.
Si on casse la barrière entre les organismes, on peut s'amuser à faire une petite expérience de pensée. On peut concaténer tous les génomes de toutes les espèces vivantes sur terre (je n'intègre pas tous les mutants pour faire simple). Partons avec une fourchette large: 100 millions d'espèces. Soyons large également sur la taille moyenne d'un génome : 1 milliard de nucléotides. On a donc une séquence ADN de 1017 nucléotides de long représentant le génome de la biosphère. Cette séquence forme un réseau gigantesque d'interaction inter-nucléotidique épistatique. Reposons nous maintenant la question pourquoi gènes et organismes nous semblent tant adaptés comme unité de sélection de base ? Car dans cet immense réseau épistatique de la biosphère, les gènes et organismes représentent des points chauds à forte densité d'interaction inter-nucléotidique. Le gène tout d'abord car on comprend bien que les interactions épistatiques inter-nucléotidiques d'un gène sont très nombreuses et très informatives. Autrement dit, la densité d'interactions inter-nucléotidiques au niveau d'un gène semble supérieure à celle d'une région non codante. Enfin l'organisme car il représente aussi peut être un point chaud très dense en interactions épistatiques. En effet, le nombre d'interactions épistatiques unissant les nucléotides d'un même génome semble supérieur au nombre d'interactions épistatiques unissant les nucléotides de génome/organisme différents. La nature pourrait ainsi trouver plus facile d'injecter de l'information en utilisant les interactions entre nucléotides d'un même génome.
Je "switch" en mode métaphysique pour anticiper sur les chapitres suivants. La vie sur terre pourrait se caractériser par cet immense réseau épistatique entre nucléotides. Imaginez maintenant le paysage adaptatif de ce super génome de 1017 nucléotides. Et posez-vous la question suivante: que mettez-vous sur l'axe Z ? Si vous mettez quelque chose, vous admettez qu'il y a un but, une finalité à la vie, à la biosphère, une téléologie, une téléonomie, une fonction qui est optimisée.

Ce que je mettrai bien sur l'axe Z, c'est la volonté de puissance13 de la biosphère. La biosphère n'a pas la volonté de vivre mais de s'étendre, de gagner en puissance. Regardez jusqu'où réussit à se loger la vie : dans les fonds marins, dans les geysers... La vie mène un combat contre le monde minéral. Elle cherche, via son algorithme évolutif, à envahir les zones qui lui résistent. La biosphère est, en l'état actuel de nos connaissances, limitée à la surface de la croûte terrestre. Nul doute qu'elle essaie par tous les moyens de gagner en profondeur. Mais il y aussi la conquête de l'univers que la vie voudrait réussir. Le cerveau humain sera-t-il l'outil qui permettra à la vie de réussir cette conquête? Ainsi la vie pourrait chercher à coloniser la matière minérale pour l'informatiser. Les atomes dans l'univers sont trop indépendants (sur le plan informatif) les uns des autres. La vie pourrait chercher à accroitre toujours plus la quantité d'information injectée dans les atomes en créant un immense réseau épistatique interatomique (ou inter-particulaire si vous préférerez). Un vulgaire caillou contient peu d'informations par rapport à un organisme vivant contenant le même nombre d'atomes. Le but de la vie ? Accroitre sans cesse la quantité d'informations (et les capacités de calcul) présentes dans les 1080 atomes de l'univers connu. Ce chapitre est maintenant terminé. Ce dernier paragraphe, très "métaphysique" et "spéculatif", offre au lecteur une excellente transition vers les chapitres sur la vie, l'information ou le futur.
Notes de bas de page
- Nietzsche, Crépuscule des idoles, Folio p. 66
- Voir l'article de Frank J. Sonleitner What Did Karl Popper Really Say About Evolution?
- Stuart Kauffman, At home in the universe, p. 165.
- Stuart Kauffman, At home in the universe, p. 30.
- Hervé Zwirn, les systèmes complexes, Odile Jacob p. 96.
- Stuart Kauffman, At home in the universe, p. 49.
- L'image est issue de Wikipedia.
- Notez que cette navigabilité du paysage adaptatif est toujours embêtante car elle se base sur ce que notre esprit peut "construire" c'est dire un paysage en 3 dimensions alors qu'il nous faudrait un esprit capable de construire cognitivement en n dimensions. Cet esprit là serait une sorte de surhomme et cette question est réservée aux chapitres sur le futur auquel je renvoie mon lecteur.
- Stuart Kauffman, At home in the universe, p. 101.
- Stuart Kauffman, At home in the universe, p. 104.
- Thomas Pradeu, Chapitre sur la Philosophie de la biologie dans Précis de philosophie des sciences.
- Photo Wikipedia du lac Morraine dans le parc national de Banff prés de Calgary au Canada.
- Concept de Friedrich Nietzsche.