Informatique et biologie
Préambule
Dans ce chapitre, nous allons étudier les liens existant entre informatique et biologie. Plus spécifiquement, je me focaliserai sur les analogies et différences entre cellule et ordinateur. Un point important qu'avait remarqué William Ross Ashby c'est, qu'avant l'arrivée des ordinateurs, il n'y avait pas de système de complexité moyenne. Un trou énorme existait entre la complexité d'un système vivant (un arbre par exemple) et le reste du monde physico-chimique. Ce trou a été comblé par l'arrivée de l'informatique qui a offert à la fois de nouveaux outils / un nouveau système complexe à étudier pour comprendre les règles de fonctionnement de systèmes encore plus complexes: les être vivants.
Des nouvelles disciplines: l'intelligence artificielle et la vie artificielle
A la différence d'un automate comme le canard de Vaucanson, qui fonctionne selon une succession d'opérations préétablies, un robot peut modifier dynamiquement son comportement. Un organe sensoriel (capteur), en donnant une information sur l'environnement extérieur, peut modifier l'activité d'un organe moteur par l'intermédiaire d'une architecture de contrôle (pseudo système nerveux).
Les premiers robots qui imitèrent la vie d'une manière vraiment impressionnante (dans les années 50) furent les tortues cybernétiques du neurophysiologiste William Grey Walter1. Ce dernier appela ses deux premières tortues Elmer et Elsie, acronymes de "Electro MEchanical Robots Light Sensitive". Leur mécanisme interne comprenait un oeil photoélectrique et deux amplificateurs à tube commandant des relais qui activaient des moteurs de direction et de progression. Les deux tortues donnaient l'impression d'explorer leur environnement. Elles repéraient les sources de lumière de faible intensité dans les environs et tournaient autour. Elles pouvaient contourner les obstacles et éviter les sources de lumière plus vives qui avaient pour effet de les "brûler". Mais lorsque que leurs batteries commençaient à faiblir, elles y puisaient en fait leur énergie. Si la lumière était fixée sur les créatures elles-mêmes, elles montraient alors des mouvements encore plus complexes d'attraction et de répulsion qui évoquaient certains comportements sociaux de défense de territoire ou de reproduction. Ces robots, en ne répétant jamais exactement la même action, montraient une sorte d'indétermination et de spontanéité rappelant le schéma général d'action d'un animal. Ce comportement émergent qui ressemble à la vie est une forme précoce de ce qu'on appelle maintenant "la vie artificielle".
La "vie artificielle" est un champ de recherche interdisciplinaire alliant informatique et biologie dont l'objectif est de créer des systèmes artificiels s'inspirant des systèmes vivants, soit sous la forme de programmes informatiques, soit sous la forme de robots2. C'est la discipline "soeur jumelle" de l'intelligence artificielle. Je développe ici quelques exemples/résultats obtenus dans ces disciplines.
Le robot Kismet, développé dans les années 90 au MIT, possède un système auditif, visuel et expressif lui permettant de participer à des interactions humaines et de démontrer en même temps sa capacité à "simuler" des émotions (Breazeal 1998). Quand un visiteur se met devant, l'expression du robot change en une expression très convaincante d'intérêt et de plaisir. Ce qui arrive après dépend bien sûr de l'attitude du visiteur: si celui-ci approche ses mains vers le robot, ce dernier semble agacé. Si le visiteur montre des couleurs vives, le robot sourit. De nombreux visiteurs rapportent qu'il est très difficile de détourner le regard quand Kismet vous fixe dans les yeux. Et il est quasiment impossible de ne pas anthropomorphiser la machine.
Il existe également des programmes de simulation de "vie artificielle" essayant de reproduire le plus fidèlement possible certaines caractéristiques de la vie (évolution, autoreproduction, adaptation à l'environnement...). Framsticks, par exemple, est un simulateur tridimensionnelle de vie artificielle (Komosinski 2009). Les organismes consistent en des structures physiques (des sortes de bâtonnets) et des structures de contrôles (pour interagir avec l'environnement ; sorte de cerveau). Ils évoluent au cours du temps en fonction du paysage adaptatif défini par l'utilisateur (la fonction à optimiser étant par exemple la vitesse) mais peuvent aussi coévoluer spontanément dans un environnement complexe.
Enfin, je ne peux résister à l'envie de vous parler de Watson, un programme informatique d'intelligence artificielle conçu par IBM dans le but de répondre à des questions formulées en langue naturelle. En Février 2011, Watson a battu les deux champions du monde (humains) au jeu télévisé américain Jeopardy. Le présentateur pose n'importe quelle question aux candidats (transcrite en texte pour le programme) et Watson "comprend" puis répond en moyenne plus vite et mieux que les candidats humains. Il n'a pourtant pas accès à Internet... mais dispose de 200 millions de page de contenu (incluant tout Wikipedia) sur 4 téraoctets de disque dur.

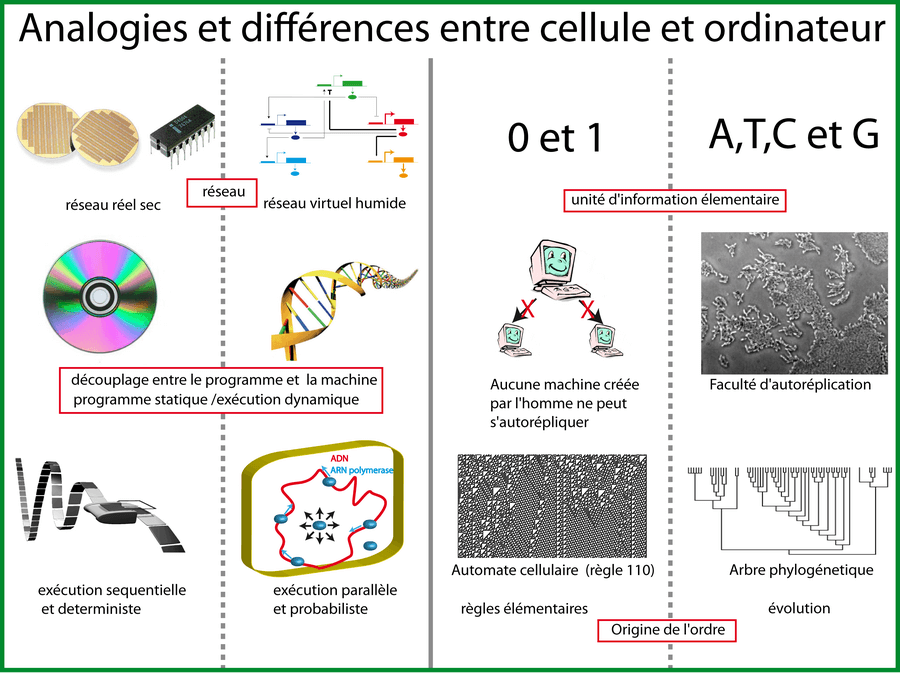
Je dirais que les trois techniques qui ont été le plus utilisées dans le développement de la vie artificielle sont les automates cellulaires (un système dynamique discret avec des règles fixes et simples mais capable de faire émerger une grande complexité), les réseaux de neurones (capables d'apprentissage et donc de fournir un mécanisme perceptif indépendant des idées du programmeur) et les algorithmes évolutionnistes (servant à résoudre des problèmes d'optimisation ; par exemple l'algorithme génétique). Dans les dénominations de ces trois techniques apparaissent des mots issus de la biologie: "cellulaires", "neurones" et "génétique". Pourquoi les informaticiens empruntent-ils du vocabulaire à la biologie pour décrire le fonctionnement de leurs algorithmes ? D'où viennent les liens entre informatique et biologie, quelles sont les analogies et différences entre une cellule et un ordinateur. Il est temps maintenant de regarder tout cela de plus prés.
Actuellement, la différence la plus évidente entre une cellule et un ordinateur, c'est la taille. Une bactérie comme E. coli fait environ 1µm de large sur 2µm de long alors que les microprocesseurs de nos ordinateurs et téléphones ont encore une taille de l'ordre du centimètre même si la miniaturisation se poursuit.
La deuxième différence c'est le fait que le réseau nécessaire au calcul dans un circuit intégré est réel alors qu'il est virtuel dans une bactérie (voir chapitre sur les interactions).
La troisième différence qui saute aux yeux c'est que la partie "hardware" de nos ordinateurs est "sèche" alors qu'une cellule est faite de matière humide "wetware".
Il existe pourtant une analogie évidente entre ordinateur et cellules: dans les deux cas, il y a découplage entre le programme et la machine. L'ordinateur et le cd-rom (ou la clef USB pour être plus moderne) sont deux entités bien distinctes. Pour ceux qui préfèrent parler de machine de Turing (modèle abstrait du fonctionnement d'un ordinateur), il y a bien un découplage entre "le ruban " et la "tête de lecture". Dans une cellule, c'est la même chose, il y a bien découplage entre la machine (machinerie globale, métabolisme: toute la cellule sauf l'ADN) et le programme c'est-à-dire l'ADN (Danchin 2008). Dans les deux cas, on a bien un programme statique (qui ne change pas) et une exécution dynamique.
Cependant cette exécution revêt des différences de poids. La machine de Turing (=l'ordinateur) avance le long du ruban (=le long du programme) de manière séquentielle alors que l'ADN d'une cellule est lu et exécuté de manière parallèle par les ARN polymérases et les ribosomes3. De plus, une machine de turing est strictement déterministe alors que la cellule "compute" de manière probabiliste via le mouvement brownien des molécules.
Les processeurs des ordinateurs sont composés de transistors ne gérant chacun que deux états. Un programme informatique est donc, au final, interprété par la machine comme une suite de 0 et de 1 (système binaire: des bits). Au contraire, l'ADN est une suite, non pas de deux états, mais de 4 états: les 4 bases nucléotidiques (A, T, C, G).
Un ordinateur est incapable de s'auto-reproduire. D'ailleurs aucune machine créée par l'homme n'a actuellement cette faculté alors qu'une cellule en est capable. Faire deux bactéries, c'est même la "finalité" (vous verrez plus loin que je n'ai pas peur de ces anthropocentrismes) d'une bactérie.
On doit à John von Neumann l'explication théorique du fonctionnement d'un système auto-réplicatif4 (Fatés 2001). Cet homme, à l'esprit décidément très créatif, a aussi suggéré de lâcher la bombe A "Fat man" sur Kyoto, idée finalement écartée par Roosevelt5:
- Un système auto-réplicatif contient une description de lui-même (dans une cellule, cette description c'est l'ADN)
- Il ne contient pas (ne nécessite pas) une description de la description (ce qui évite la récursivité à l'infini) (il n'y a pas besoin dans l'ADN d'une partie qui décrit l'ADN)
- La description (l'ADN) est utilisée deux fois, comme ensemble d'instruction (transcription et traduction), et comme support qui peut être copié (réplication)
- Un constructeur universelle (ARN polymérases + ribosomes) est capable d'interpréter la description en construisant un grand nombre d'objets (les protéines)
- Un des objets (l'ADN polymérase) construit par le constructeur universel est capable de copier la description.
Revenons maintenant à nos différences entre cellule et ordinateur: il y a une différence essentielle relative à la notion d'ordre et de complexité. En biologie, on vit dans le "paradigme de l'évolution": toute structure complexe, contenant de l'ordre (une cellule, un oeil, une girafe, une enzyme) ne peut provenir que de l'algorithme génétique (l'évolution) qui "tourne " depuis quelques milliards d'années en générant des mutations et des recombinaisons puis sélectionne inexorablement les structures les mieux adaptées.
A l'inverse, en informatique, des structures ordonnées, désordonnées ou à la frontière entre "ordre et chaos" peuvent émerger à partir de règles très simples, élémentaires même (c'est-à-dire sans nécessiter 4 milliards d'années d'évolution). C'est ce que l'on observe avec les automates cellulaires que nous allons étudier maintenant.
Les automates cellulaires
Un automate cellulaire6 est un système dynamique discret consistant en une grille régulière où chaque case de la grille (appelée cellule) peut prendre un ensemble fini d'états (par exemple 2 états: noir ou blanc). Le système peut évoluer au cours du temps: l'état d'une cellule au temps t+1 est fonction de l'état au temps t d'un nombre fini de cellules appelé son "voisinage". À chaque nouvelle unité de temps, les mêmes règles sont appliquées simultanément à l'ensemble des cellules de la grille, produisant une nouvelle "génération" de cellules qui dépendent entièrement de la génération précédente.
L'automate cellulaire non trivial le plus simple que l'on puisse concevoir consiste en une grille unidimensionnelle de cellules ne pouvant prendre que deux états ("0" ou "1"), avec un voisinage constitué, pour chaque cellule, d'elle-même et des deux cellules qui lui sont adjacentes. Chacune des cellules pouvant prendre deux états, il existe 23=8 configurations (ou motifs) possibles d'un tel voisinage. Pour que l'automate cellulaire fonctionne, il faut définir quel doit être l'état, à la génération suivante, d'une cellule pour chacun de ces motifs. Il y a 28=256 façons différentes de s'y prendre, soit donc 256 automates cellulaires différents de ce type.
Les automates de cette famille sont dits "élémentaires". On les désigne souvent par un entier entre 0 et 255 dont la représentation binaire est la suite des états pris par l'automate sur les motifs successifs 111, 110, 101, etc.
À titre d'exemple, considérons l'automate cellulaire défini par la table suivante, qui donne la règle d'évolution suivante:
Cette table montre que si par exemple, à un temps t donné, une cellule est à l'état "1", sa voisine de gauche à l'état "1" et sa voisine de droite à l'état "0" (3ème colonne du tableau), au temps t+1, la cellule centrale sera à l'état "0". Par convention, la règle présentée dans le tableau ci-dessus est nommée "règle 30", car 30 s'écrit 00011110 en binaire et 00011110 correspond à la deuxième ligne du tableau ci-dessus, décrivant la règle d'évolution.
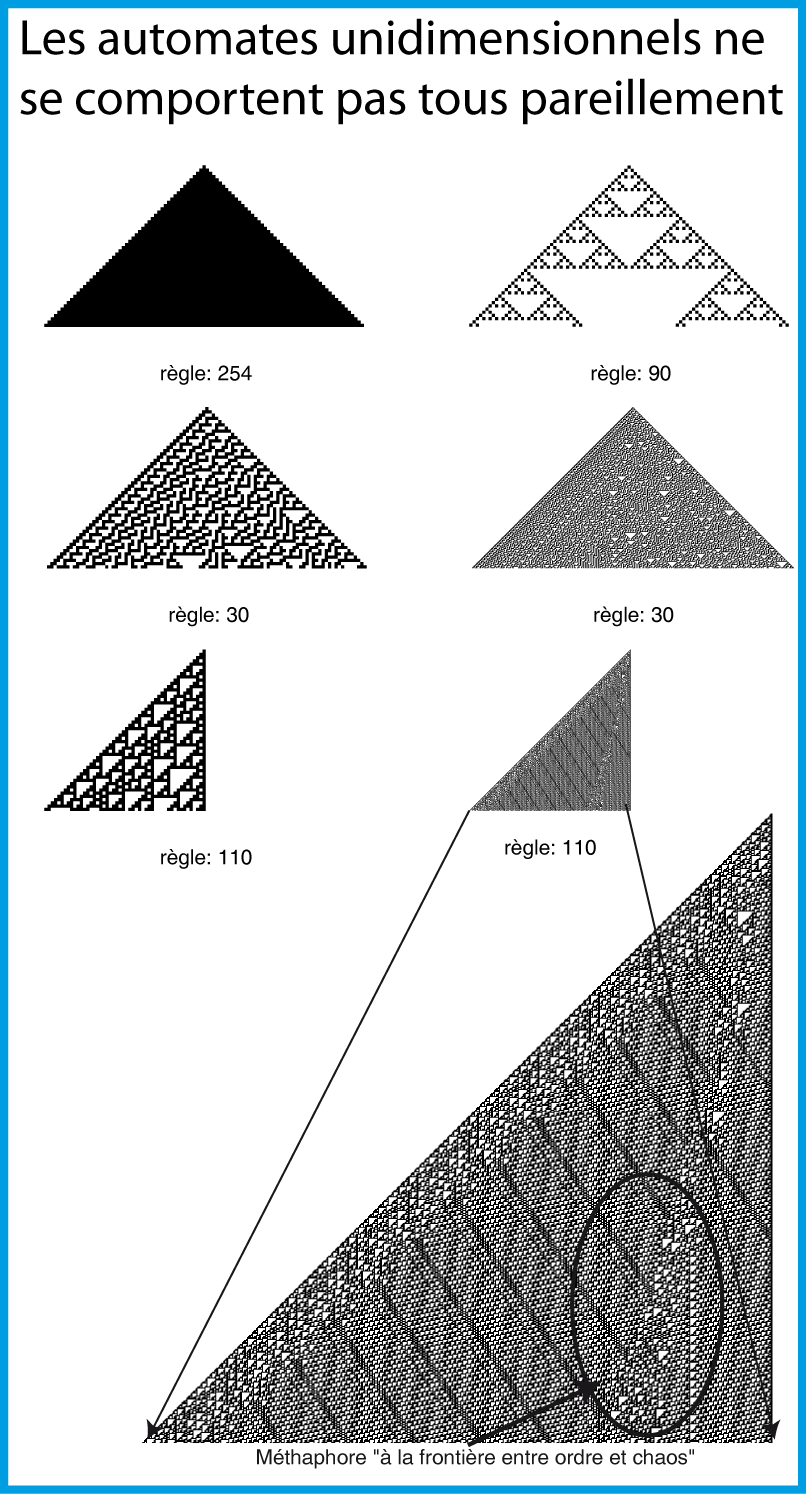
Dans un automate élémentaire unidimensionnel, le temps est représenté du haut vers le bas c'est-à-dire que chaque ligne est le résultat de la ligne précédente. Si l'on part d'une grille initiale où toutes les cellules sont à l'état "0" sauf une et que l'on suit la "règle 30 " on aboutit au résultat visible sur la figure 3.
Les 256 règles (pourtant très proches) ne conduisent pas toutes au même genre de structures (j'ose le mot "phénotype"). Stephen Wolfram (Mathématicien célèbre à qui l'on doit notamment, le logiciel de calcul formel Mathematica) a proposé une classification de ces structures (Wolfram 1984):
- Classe I: presque toute configuration initiale conduit à un état homogène (exemple avec la règle 254 sur la figure 3).
- Classe II: des structures stables ou périodiques émergent, mais rien de plus (exemple avec la règle 90 sur la figure 3).
- Classe III: comportement chaotique avec des motifs apériodiques. À long terme les fréquences d'apparitions des différents motifs se stabilisent (exemple avec la règle 30 sur la figure 3)
- Classe IV: "émergence" de structures complexes capables d'osciller, de se mouvoir, voire de persévérer plus ou moins dans leur auto-organisation malgré des perturbations structurelles (exemple avec la fameuse règle 110 sur la figure 3)
C'est cette dernière classe qui est particulièrement intéressante pour nous biologistes. En effet, l'émergence (visuelle) de ces structures complexes ne peut manquer de nous rappeler un phénomène vivant: par exemple la zone que j'ai entourée d'un cercle sur la règle 110 (figure 3) nous rappelle la caractéristique fondamentale qu'attribue Stuart Kauffman à la vie qui nagerait "à la frontière entre ordre et chaos".
Ce qui est "génial" avec les automates cellulaires, c'est qu'ils sont, pour nous biologistes, une transition douce, didactique, presque expérimental vers l'informatique et les mathématiques. La figure 3 est générée par un script Matlab de 30 lignes seulement. J'indique au début du script les numéros des règles que je veux voir "plotter" (254, 90, 30 et 110) et le script se charge de faire les calculs (les plus téméraires se frotteront aux 2 lignes représentant le coeur de l'algorithme emprunté à la technique/l'astuce de David Young).
Cliquer pour voir le code Matlab
%code pour générer 6 petites figures d'automates cellulaires élementaires
figure('Color', 'w');
Regles=[254 90 30 30 110 110];% regles des automates cellulaires
NombreLignes=[50 50 50 200 50 500];
NombreColonnes=[100 100 100 400 100 1000];
for j=1:6
Regle=Regles(j);
NombreLigne=NombreLignes(j);% nombre total de ligne
NombreColonne=NombreColonnes(j);% nombre total de colonne
% J'utilise le booleen 1(off) et 2(on) et non 0 et 1
Ligne=ones(1, NombreColonne);% Ligne en cour d'analyse
Ligne(floor((NombreColonne+1)/2))=2;% cellule du milieu est "on" (2)
RegleCorrespondante = (bitget(Regle, 1:8) + 1);% decompresse les règles
Grille= ones(NombreLigne, NombreColonne);% allocation pour la grille
% Iteration pour generer le reste de la grille
for i=1:NombreLigne
Grille(i, :) = Ligne;% Ecrit l'état courant dans la ligne en cour
% étapes centrales : applique les règles des automates cellulaires à
%à propager le long du patterne 1D
% J'utilise la technique de David young: les deux lignes ci-dessous ne
% sont pas simples à comprendre...prendre son temps...
indice = sub2ind([2 2 2], ...
[Ligne(2:end) Ligne(1)], Ligne, [Ligne(end) Ligne(1:end-1)]);
Ligne = RegleCorrespondante(indice);
end
subplot(3,2,j);
% repasse en binaire classique (0et1)puis inverse les couleurs
imshow(~(Grille-1));
xlabel(['règle: ' int2str(Regle)])
end
Précisons tout de même que derrière l'apparente simplicité expérimentale des automates cellulaires se cachent des problèmes de mathématiques formels bien plus difficiles (1000 fois trop difficile pour moi d'ailleurs). Mais je le répète, pour nous biologistes, la seule chose qui importe c'est de constater visuellement, "de prendre acte" que des structures complexes peuvent émerger à partir de règles triviales. Rien à voir avec les milliards d'années d'évolution nécessaires chez les être vivants. Et c'est cela qui ne peut manquer de questionner (à tort ou à raison) tout biologiste "honnête avec lui même" sur la valeur du paradigme évolutionniste et ses alternative possibles.
Le jeu de la vie de Conway
Le jeu de la vie (attention il ne s'agit pas d'un jeu en réalité) a été imaginé par John Conway dans les années 70. C'est probablement l'un des automates cellulaires les plus connus. Malgré des règles très simples, le jeu de la vie génère des motifs complexes. Le jeu se déroule sur une grille à deux dimensions dont les cases appelées cellules peuvent prendre deux états distincts: vivantes ou mortes. Comme précédemment, à chaque itération, on calcule l'évolution de chacune des cellules de la grille mais cette fois-ci, l'automate est bidimensionnel: l'évolution d'une cellule est entièrement déterminée, non pas comme tout à l'heure par ses deux voisines, mais par l'état de ses huit voisines. Le temps n'est plus représenté du haut vers le bas par des lignes représentant les itérations successives mais par des trames, à la manière d'un film, représentant l'état de la grille à chaque itération.
Rendez-vous compte: l'automate du jeu de la vie ne suit que les deux règles suivantes:
- Une cellule morte qui possède exactement trois voisines vivantes devient vivante (elle naît).
- Une cellule vivante qui possède deux ou trois voisines vivantes le reste, sinon elle meurt.
Ainsi, ces deux règles suffisent à générer des patterns complexes (Figure 4). La concision du script Matlab dénote par rapport à la complexité des patterns observés sur la vidéo générée par le script. Encore une fois, je souhaite vous faire remarquer que notre oeil puis notre cerveau attribue à ces patterns une sorte de pseudo-activité vivante. D'où l'analogie "cellule" et le nom donné à cet automate "jeu de la vie".
Cliquer pour voir le code Matlab
%Jeux de la vie de conway
% si le script génére un erreur, commenter le code du film
clear all
F=figure('color','w');
%mov = avifile('conway4.avi','quality',100,'compression','none');% code film
n=200; % taille de la grille (carré de longueur n)
%taille de la matrice
cellule=(rand(n,n)>0.7);% matrice generée avec quelques cellules "on" (1)
somme=zeros(n,n);% alloue l'espace à la variable somme
x = 2:n-1;
y = 2:n-1;
for i=1:200
%somme des voisins les plus proches
somme(x,y) = cellule(x,y-1) + cellule(x,y+1) + ...
cellule(x-1, y) + cellule(x+1,y) + ...
cellule(x-1,y-1) + cellule(x-1,y+1) + ...
cellule(3:n,y-1) + cellule(x+1,y+1);
% regle du jeux de la vie
cellule = (somme==3) | (somme==2 & cellule);
%montre les images à chaque iteration et inverse les couleurs
imshow(~cellule)
%mov = addframe(mov,F);% code film
pause(0.1)
end
%mov = close(mov);% code film
Il me semble que c'est cette simple analogie visuelle, ce regard porté à cette complexité émergente, qui est l'unique raison unissant automate cellulaire (Mathématique, informatique) et biologie. Je souhaite faire ici une parenthèse sur cette question des analogies, des métaphores. J'ai trouvé assez souvent, dans des ouvrages de scientifiques ou de philosophes, une critique forte vis-à-vis de ces analogies par exemple chez Bachelard:
"Aussi l'esprit scientifique doit-il sans cesse lutter contre les images, contres les analogies, contre les métaphores" 7
Je pourrais facilement sombrer dans le positivisme dogmatique et m'élever formellement contre le lien visuel invérifiable, infalsifiable, ce lien si mince qui relie "l'impression de vie" avec "l'automate cellulaire de Conway". Mais je soupçonne au contraire ces analogies de cacher plus qu'elles ne laissent transparaitre. Pour prendre un autre exemple, j'ai remarqué qu'en biologie, on utilise beaucoup d'images "humaines": les gènes "égoïstes", un phénomène "altruiste", la "communication" intercellulaire, le "code" ou " texte" génétique, "la fonction" ou " finalité" d'une enzyme, d'un organe. L'interprétation la plus classique de ces analogies c'est l'anthropocentrisme: la fâcheuse tendance à tout ramener à l'homme. Or, il me semble que "ces dérives" des analogies existent principalement en biologie, beaucoup moins en physique ou en chimie par exemple. La question qui vient alors immédiatement à l'esprit c'est: pourquoi en biologie, les scientifiques trouvent autant de "concepts" à dimension humaine ? Deux réponses possibles: la faute anthropocentrique ou, comme je le soupçonne, un lien indicible, complexe, indiscernable mais qui donne à ces concepts (probablement à redéfinir) une dimension plus universelle dans le règne vivant qu'on ne l'imagine a priori.
Ainsi, je ne condamnerais pas les métaphores et les images aussi vite. Elles sont souvent infalsifiables et irrationnelles mais, selon moi, elles restent une pièce cruciale (par exemple les "fous") de l'échiquier scientifique. Elles construisent et orientent la pensée. Parfois en mal mais le plus souvent, je le soupçonne, en bien.
Un peu de grammaire (les systèmes de substitution séquentielle)
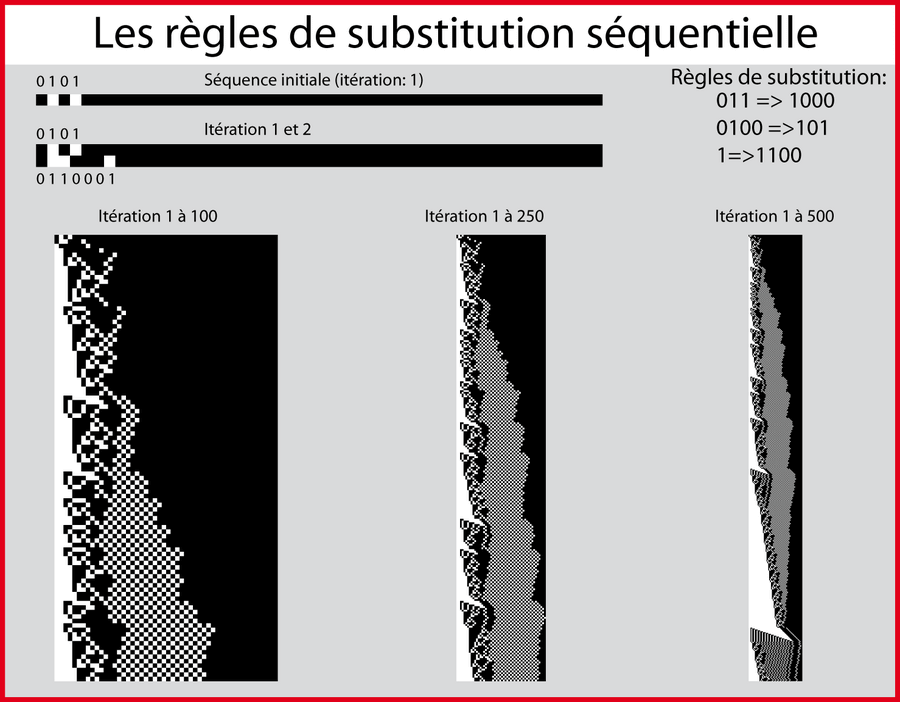
Les automates cellulaires ne sont pas seuls à pouvoir créer de la complexité à partir de règles simples. Stephen Wolfram identifie, dans son ouvrage A new kind of science, un grand nombre de systèmes montrant des caractéristiques similaires. Je vais en détailler un en particulier: le système de substitution séquentielle8. Derrière ces mots barbares se cache ni plus ni moins que la fonction "find and replace" de votre éditeur de texte préféré. Concrètement, je définis initialement 3 règles de substitution (de grammaire):
- Si l'algorithme trouve le motif 011, il le remplace par le motif 1000.
- Si l'algorithme trouve le motif 0100, il le remplace par le motif 101.
- Si l'algorithme trouve le motif 1, il le remplace par le motif 1100.
Puis l'algorithme scanne la séquence initiale de gauche à droite, opère le remplacement selon les règles de substitution puis passe à l'itération suivante. La visualisation des itérations s'effectue, comme avec les automates élémentaires, avec des lignes en noir et blanc (0 et 1) de telle sorte que la nouvelle ligne soit le résultat de la substitution effectuée sur la ligne précédente (figure 5). En fonction de règles, on peut, comme avec les automates, générer des patterns homogènes, réguliers, aléatoires ou un peu "bizarres" comme le pattern présenté sur la figure 5.
Cliquer pour voir le code Matlab
% système de substitution sequentielle
% ce script tente de reproduire le systeme de la figure (h) page 91 de
% l'ouvrage de Stephen Wolfram "A new kind of science".
% Ce systeme fonctionne comme la fonction "find and replace" des editeurs
% de texte. Dans la Sequence de base S, l'algorithme trouve la sequence
% 011, il la remplace par 1000. Idem avec 0100 =>101 et 1=>1100
%le script fait evoluer ce systeme au cours du temps et chaque ligne
%represente une itération successive.
%evidemment, les 0 et 1 sont remplacés dans l'image par noir et blanc.
clear all
% grammaire (règle de substitution)
Sequence{1}='0 1 1';% si trouve 1, remplace par 6
Sequence{2}='0 1 0 0';% si trouve 2, remplace par 7
Sequence{3}='1';% si trouve 3, remplace par 8
Sequence{6}='1 0 0 0';
Sequence{7}='1 0 1';
Sequence{8}='1 1 0 0';
% Sequence initial qui evolue au cour du temps en fonction de la grammaire
S='0 1 0 1';
Matrice=zeros(500,50);% alloue la memoire pour l'image correspondante
for iteration=1:500
% écrit S dans la ligne numero "iteration".
Matrice(iteration,1:length(str2num(S)))=str2num(S);
for i=1:3 % cherche la sequence 1, puis 2 puis 3 dans S
Match=findstr(Sequence{i},S);
% si il trouve la sequence, il opère la substitution
if ~isempty(Match)
S1=S(1:Match(1)-1);
S2=S(Match(1)+length(Sequence{i}):length(S));
S3=Sequence{i+5};
S=[S1 S3 S2];
break
end
end
end
% montre l'image du résultat
h=figure
imshow(Matrice)
saveas(h,'Grammaire500','eps')% sauve l'image en format vectoriel
Mais vous allez me dire: oui mais en quoi cela est-il intéressant ? N'est-ce pas un peu redondant avec la partie sur les automates cellulaires élémentaires ? J'aime cet exemple car il nous permet de dresser un pont vers les travaux de Stuart Kauffman. Ce dernier a écrit deux ouvrages The origin of order et son équivalent un peu simplifié (parfaitement suffisant pour notre objectif) nommé At home in the universe.
L'hypothèse principale des ouvrages de Stuart Kauffman est la suivante: de l'ordre peut émerger sans forcement que cela soit dû à l'évolution. La vie se situerait et évoluerait à la frontière entre ordre et chaos. Cette frontière ne serait pas nécessairement le produit de l'évolution. Au contraire, elle lui serait "antérieure", elle la "précéderait", elle serait un concept "de plus bas niveau" si vous préférez.
Pour argumenter l'émergence de l'ordre en absence de toute pression de sélection, Stuart Kauffman développe un certain nombre d'idées (voir chapitre sur l'évolution II). Je vais en développer une ici9. L'expérience qui suit est à connecter au modèle de "la soupe originelle" expliquant les origines de la vie.
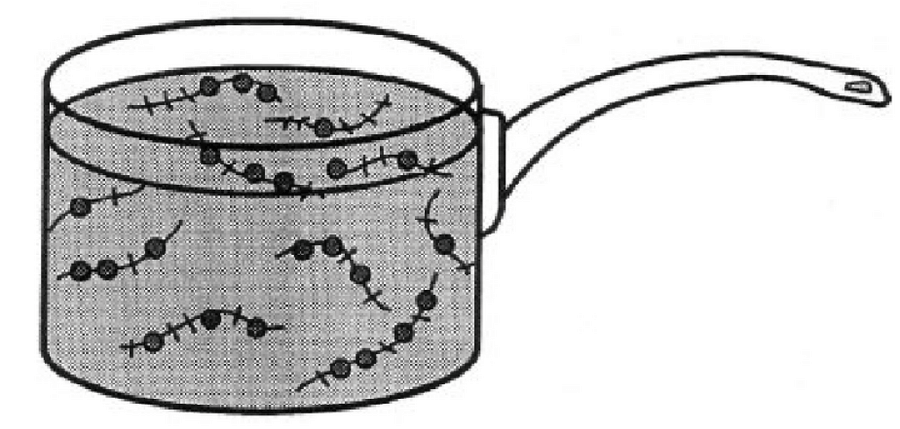
Prenez une casserole et ajoutez-y vos différentes séquences de bits (suite de 0 et de 1). Ces séquences doivent être envisagées comme des molécules. Comme tout à l'heure, il existe des règles de substitution mais cette fois-ci, celles-ci fonctionnent en trio. Si par exemple un substrat (par exemple 111) rencontre une enzyme (01001) alors on substitue le substrat (111) par le produit (010).
Tirez maintenant deux de ces séquences (molécules) au hasard (on imagine qu'il s'agit d'une collision): si vous tirez un substrat et son enzyme correspondante alors remplacez dans la soupe le substrat par le produit, puis recommencez depuis le début: tirez deux séquences (molécules) au hasard, s'il y a correspondance avec une règle de grammaire, opérez la substitution et ainsi de suite. Au fur et mesure, de nouveaux motifs (séquences) vont être créés. Ces motifs sont autant de nouveaux sites enzymatiques potentiels capables de reconnaitre un nouveau substrat pour le convertir en nouveau produit. Et c'est tout ce qu'il faut: on a une chimie algorithmique spécifiée par une grammaire spécifique. Les séquences de bits dans une casserole se transforment en nouvelles séquences de bits et ce à l'infini générant une "floraison" de nouvelles séquences. C'est l'émergence de ces patterns de floraison au cours du temps qui est intéressante. Car de là peut naitre de l'ordre mais également des structures d'une grande complexité: selon Stuart Kauffman, ce modèle de soupe originelle pourrait expliquer ni plus ni moins que la coévolution technologique de nos sociétés ! Il ne s'agit bien sûr que de simples métaphores/spéculations mais alors pourquoi nous semblent-elles taper si justes ? Pourquoi nous semblent-elles si "orientantes" ?

- Longueur de la séquence (soupe de molécules): 50 bits (gardée constante)
- Nombre de règles de substitution (grammaire): 150 (définies aléatoirement)
- Taille maximum des enzymes, substrats et produits: 15 bit
- Taille minimum des enzymes, substrats et produits: 3 bits
- Nombre d'itérations: 1500
Pour faciliter la visualisation des résultats (éviter des colonnes trop longues de 1500 lignes), les colonnes sont poolées en carré (par tranche de 250 lignes) comme indiqué sur le schéma en haut à gauche. L'encadré rouge sur le carré du milieu montre 10 lignes de 50 bits soit les 10 premières itérations. Les ronds rouges sur les carrés du bas montrent des zones qui attirent l'oeil: représentent-ils quelque chose ayant des similitudes avec la vie ? Voir le code Matlab ci-dessous qui gènère cette figure.
Cliquer pour voir le code Matlab
% petit script pour tester l' analogie entre grammaire et soupe originelle
clear all
F=figure('color','white');
NombreIteration=1500;
LongueurChaine=50;
NombreRegle=150;
Diviseur=6;%sert à pooler les iterations dans une images
NombreImages=50;% nombre de fois que la simulation tournera.
TailleMaxPerm=15;
TailleMinPerm=3;
% test l'algorithme un certain nombre de fois avec des grammaires
% differentes
for t=1:NombreImages
for i=1:NombreRegle
% pour chaque regle, decide de la taille du string à remplacer
Taille=randi(TailleMaxPerm-TailleMinPerm)+TailleMinPerm;%
% choisit aleatoirement un string subtrat, puis enzyme, puis produit
Sequence{i}.substrat=num2str(round(rand(1,Taille)),'%1d ');
Sequence{i}.enzyme=num2str(round(rand(1,Taille)),'%1d ');
Sequence{i}.produit=num2str(round(rand(1,Taille)),'%1d ');
end
%String de depart
S=num2str(round(rand(1,LongueurChaine)),'%1d ');
%alloue la taille de la grille image
Grille=false(NombreIteration/Diviseur,LongueurChaine*Diviseur);
for j=1:NombreIteration
%cette ligne un peu complexe sert à pooler les iterations en colonne de la
%largeur de LongueurChaine et de la longueur de NombreIteration/diviseur
% cela permet de visualiser un grand nombre d'iteration dans une seule
% grande image.
Grille((mod((j-1),NombreIteration/Diviseur)+1),(LongueurChaine*...
ceil(j/(NombreIteration/Diviseur))-LongueurChaine+1)...
:(LongueurChaine*ceil(j/(NombreIteration/Diviseur))))=str2num(S);
%applique l'ensemble des regles à chaque iteration mais dans un ordre
%aleatoire
for i=randperm(NombreRegle)
% cherche la sequence du substrat dans la sequence S
MatchSubstrat=findstr(Sequence{i}.substrat,S);
% cherche la sequence de l'enzyme dans la sequence S
MatchEnzyme=findstr(Sequence{i}.enzyme,S);
% si l'enzyme et le substrat sont présent alors...
if ~isempty(MatchSubstrat) && ~isempty(MatchEnzyme)...
&& length(S)>length(Sequence{i}.substrat)+1
% fait autant de remplacement que l'element (enzyme ou
% substrat) limitant
for z=1:min([length(MatchSubstrat) length(MatchEnzyme)])
%remplacement aleatoire
Var=randperm(length(MatchSubstrat));
% change le substrat par le produit
S1=S(1:MatchSubstrat(Var(z))-1);
S2=S(MatchSubstrat(Var(z))+...
length(Sequence{i}.substrat):length(S));
S3=Sequence{i}.produit;
S=[S1 S3 S2];
end
end
end
end
imshow(Grille)
saveas(F,num2str(t),'png')%sauve chaque image avec son numero
%pause(0.1)
end
A partir de la description de Stuart Kauffman de la chimie algorithmique, je n'ai pas vraiment réussi à comprendre si sa description correspondait à une simple argumentation/métaphore ou si celle-ci était confortée par des résultats de simulation. En effet, l'auteur rentre ensuite dans des considérations mathématiques abstraites et complexes qui me dépassent totalement. Biologiste de formation, j'ai souhaité resté à un niveau que je qualifierais de plus expérimentale c'est à dire plus à ma portée: j'ai essayé d'encoder la soupe originelle de séquences sous Matlab. Il faut considérer cela comme un jeu dont on sait, à la base, qu'il n'en sortira rien d'autre que "l'aspect formateur" de la démarche.
Cela a été plus compliqué que je m'y attendais. Partant de la description de Kauffman, il a néanmoins fallu que j'opère un certain nombre de choix qui, au final, m'ont peut être fondamentalement éloigné de ce qu'avait en tête son auteur.
Je ne vais pas détailler exactement la manière dont je m'y suis pris, ce serait trop long. Brièvement, une séquence de 50 bits évolue au cours du temps en fonction de 150 règles de substitution. Ces règles définissent des enzymes, des produits et des substrats qui sont des séquences en bits choisis aléatoirement. Les tailles de ces séquences sont également choisies aléatoirement mais elles sont comprises entre 3 et 15 bits. A chaque itération, toutes les règles de grammaire sont appliquées à la séquence dans un ordre aléatoire et de manière dose dépendante: si un substrat et une enzyme particulière sont présents en de nombreux exemplaires, il y aura beaucoup de produits dans la séquence suivante. J'ai fait tourner l'algorithme sur 1500 itérations ce qui représente en réalité plusieurs dizaines de milliers de substitution.
J'identifie deux questions fondamentales à se poser quand on entreprend d'encoder ce type de simulation:
- quelle est la meilleure forme de représentation des résultats ? je n'ai pas eu d'autres idées que les images (que le cerveau analyse en moins d'une seconde). La première ligne correspond à la condition initiale aléatoire: c'est une ligne de 50 bits représentées en noir et blanc (les 0 et les 1). La ligne directement en dessous est le résultat d'une itération comportant plusieurs substitutions. Les 1500 itérations forment une colonne trop longue pour être visualisée facilement (50 pixels x 1500 pixels). Dans la figure 6 illustrant les résultats obtenus lors des simulations, j'ai donc poolé des colonnes de 250 lignes ensembles ce qui génère un carré. L'angle gauche du haut de mon carré représente la première itération alors que l'angle droit du bas représente la dernière itération. Ce type de représentation et d'analyse des résultats est limité car bien que l'oeil soit extrêmement performant pour détecter des structures complexes, rappelant la vie, il ne serait en revanche pas très prudent de lui accorder une confiance excessive.
- Car: que cherche-t-on vraiment au final ? La première chose à constater en regardant les différents carrés de la figure 6, c'est la grande variabilité visuelle des images obtenues (j'en ai regardé presque mille soit plusieurs heures de simulations). Rappelons la présence de 150 règles de grammaire dans toutes les simulations. Intuitivement, on aurait pu s'attendre à ce qu'un nombre si important de règles choisies aléatoirement limite mécaniquement / statistiquement la variabilité visuelle des images. Or ce n'est pas du tout le cas: on voit que:
- de nombreuses images semblent grossièrement aléatoires (pseudo-aléatoire car la présence de la grammaire introduit un biais). Cette "aléatoire ressenti" varie dans la prédominance du blanc ou du noir et dans ce que j'appellerai la "granularité" (figure 6) : les deux carrés du haut).
- de nombreuses images finissent par atteindre un état d'équilibre (figure 6) : les deux carrés du milieu)
- certaines images, plus rares, semblent plus complexes, plus "organisées". Ce sont ces images qui rappellent le plus la vie grâce à la métaphore "la vie navigue à la frontière entre ordre et chaos".
Mais on constate aussi qu'il est difficile d'aller plus loin, de chercher des conclusions et pistes plus en profondeur. Il semble que cette approche expérimentale nous cantonne, quoi qu'il arrive, au domaine de la métaphore. Ce que nous recherchons, en réalité, dans les images, c'est une information de valeur10. Or nous ne trouvons cette information ni dans les images aléatoires, ni dans les images homogènes. Nous sommes maintenant prêts à aborder le chapitre sur l'information.
Bibliographie
- Breazeal, C. (1998). "Early Experiments using Motivations to Regulate Human-Robot Interaction".
- Danchin, A. (2008). "Bacteria as computers making computers." FEMS Microbiol Rev.
- Fatés, N. (2001). "LES AUTOMATES CELLULAIRES : VERS UNE NOUVELLE EPISTEMOLOGIE ?"
- Komosinski, M. U., S (2009). "Framsticks: Creating and Understanding Complexity of Life."
- Wolfram, S. (1984). "Universality and Complexity in Cellular Automata " Physica D.
Notes de bas de page
- Mes sources pour ce paragraphe sont: l'ouvrage de Jean-Claude Heudin, Créatures artificielles, Odile Jacob p. 170 ainsi que Wikipedia: Tortues de Bristol.
- Article de Wikipedia: Vie artificielle.
- Cette manière de voir est partiellement fausse si on imagine le travail de plusieurs "coeurs" d'un processeur ou, à l'inverse, le travail d'une seule ARN polymérase dans la cellule
- Les points ci-dessous sont repris et modifiés à partir du très bon rapport de DEA de Nazim Fatés.
- Article Wikipedia: John von Neumann.
- Cette introduction sur les automates cellulaires est très largementinspiré de l'article "automates cellulaires" de Wikipedia. Le lecteur doit donc attribuer la paternité de certains paragraphes ci-dessous aux contributeurs Wikipedia. Notez cependant que ces découvertes sur les automates cellulaires élémentaires doivent être attribuées, quant à elles, à Stephen Wolfram (A new kind of science).
- Bachelard, La formation de l'esprit scientifique, Vrin p. 45.
- Stephen Wolfram, A new kind of science, p.92.
- Stuart Kauffman, At home in the universe, P. 156-157.
- Terme emprunté à Antoine Danchin.